Bestimmung des Weizenanteils in Dinkelprodukten mittels digitaler PCR
Abstract
Seit 2021 wird der Weizenanteil in Dinkelerzeugnissen am LGL molekularbiologisch mittels digitaler PCR bestimmt. Diese in Zusammenarbeit mit dem Chemischen und Veterinäruntersuchungsamt (CVUA) Freiburg und dem Kantonalen Labor Zürich entwickelte Methode erlaubt trotz der sehr nahen genetischen Verwandtschaft von Dinkel und Weizen eine eindeutige Differenzierung und Quantifizierung des Weizenanteils. Im Jahr 2021 wurde diese Methode mit Validierungsdaten der drei beteiligten Institutionen publiziert [1]. Die laborübergreifende Validierung der Methode erfolgte 2022 in einem Ringversuch der § 64 LFGB Arbeitsgruppe „Molekularbiologische Methoden zur Pflanzen- und Tierartendifferenzierung“. Die Ergebnisse dieses Ringversuchs werden für 2023 erwartet. Das LGL untersuchte in den Jahren 2021 und 2022 insgesamt 80 Dinkelerzeugnisse (Backwaren, Teigwaren, Mehle, Dinkelkörner), zwei Proben Dinkelteigwaren wurden wegen Weizenbeimischungen beanstandet. Zwei weitere Erzeugnisse (eine Teigware, eine Probe Dinkelkörner) wurden wegen Kennzeichnungsmängeln und Werbeaussagen beanstandet, nicht jedoch wegen Weizenbeimischungen.
Hintergrund der Untersuchungen
In den Leitsätzen für Brot und Kleingebäck [2] ist festgelegt, dass Brot und Kleingebäck, in dessen Bezeichnung eine Getreideart genannt ist, diese Getreideart zu mindestens 90 % enthalten muss. So muss z. B. ein Dinkelbrot zu mindestens 90 % aus Dinkel hergestellt sein. Dinkelteigwaren enthalten nach den Leitsätzen für Teigwaren ausschließlich die namensgebende Getreideart [3].
Da in den Mühlen meist sowohl Weizen als auch Dinkel vermahlen wird, sind Dinkelmahlerzeugnisse anfällig für produktionsbedingte Beimischungen. Aufgrund des deutlichen Preisunterschiedes zwischen Dinkel und Weizen sind zudem Verfälschungen bei Dinkelprodukten nicht auszuschließen.
Molekularbiologische Analytik
Die molekularbiologische Unterscheidung von Dinkel und Weizen ist aufgrund der nahen Verwandtschaft und der damit verbundenen hohen genetischen Übereinstimmung schwierig. Für den Nachweis und die Quantifizierung des Weizenanteils in Dinkelprodukten wurde bisher die Methode der real-time PCR (qPCR) eingesetzt. Dabei sind zwei Nachweise erforderlich:
- Bestimmung der Gesamtmenge an Weizen- und Dinkel-DNA
- Bestimmung der Gesamtmenge an Weizen-DNA
Die Ergebnisse werden dann zur Bestimmung des Weizenanteils zueinander ins Verhältnis gesetzt. Obwohl diese qPCR-Methode prinzipiell gut funktioniert, hat sie jedoch auch entscheidende Nachteile. So werden für die quantitative Bestimmung gut charakterisierte und möglichst zertifizierte Referenzmaterialien benötigt, deren genaue Konzentration bekannt ist. Ein weiteres Problem besteht darin, dass nicht in allen Fällen eine spezifische Unterscheidung zwischen Dinkel und Weizen möglich ist. Die qPCR-Methode basiert auf der Arbeit von Mayer et al. (2012; [4]) und nutzt einen kleinen Unterschied in der DNA-Sequenz zwischen Weizen und Dinkel aus. Dem Dinkel fehlen im γ-gliadin-Gen an einer bestimmten Position 9 DNA-Bausteine, wodurch eine Unterscheidung möglich wird. Problematisch hierbei ist, dass es bestimmte Dinkelsorten gibt (z. B. Badenstern, Alkor oder Schwabenspelz), in die Weizen eingekreuzt wurde, wodurch sie diesen Sequenzunterschied wieder verloren haben und daher in der qPCR wie Weizen reagieren. In diesen Fällen wird der Weizenanteil im Messergebnis überschätzt.
Um diesen Problemen zu begegnen, haben das LGL, das Chemische und Veterinäruntersuchungsamt (CVUA) Freiburg und das Kantonale Labor Zürich in einem Kooperationsprojekt eine neue Methode zur quantitativen Bestimmung des Weizenanteils in Dinkelprodukten entwickelt. Diese basiert auf der digitalen PCR (dPCR) und wurde in einer 2021 publizierten Laborvergleichsstudie der drei Laboratorien validiert [1]. Die dPCR nutzt das gleiche Grundprinzip wie die qPCR, jedoch wird die zu untersuchende DNA nicht in einer Reaktion, sondern in sehr vielen Unterreaktionen gemessen. Bei der am LGL eingesetzten digital droplet PCR (ddPCR) wird die DNA auf bis zu 20.000 Tröpfchen verteilt. In jedem dieser Tröpfchen läuft nun eine separate PCR-Reaktion ab. Ist die gesuchte Ziel-DNA (hier Weizen-DNA bzw. Dinkel-DNA) in einem Tröpfchen vorhanden, kann die PCR in diesem Tröpfchen ablaufen. Dies führt zu einer erhöhten Fluoreszenz des Tröpfchens. Ist die gesuchte DNA nicht vorhanden, kommt es zu keiner Fluoreszenzerhöhung des Tröpfchens. Nach der PCR werden die Tröpfchen gezählt und die Fluoreszenz gemessen. Das Ergebnis wird als ein „0-1“-Ergebnis dargestellt, wobei „0“ bedeutet, dass die gesuchte DNA im Tröpfchen nicht vorhanden ist und „1“ bedeutet, dass die gesuchte DNA vorhanden war und das Tröpfchen daher stärker fluoresziert. Aufgrund dieses „0-1“-Ergebnisses wird diese Technik auch als „digitale PCR“ bezeichnet. Das Verhältnis der Tröpfchen mit („1“) und ohne („0“) nachzuweisender DNA hängt von der Ausgangskonzentration der Probe ab. Ist die Ausgangskonzentration hoch, sind mehr Tröpfchen positiv und weniger Tröpfchen negativ. Daher kann die Ausgangskonzentration aus dem Verhältnis von positiven zu negativen Tröpfchen, dem Tröpfchenvolumen und einer statistischen Korrektur (Poisson-Korrektur) berechnet werden.
Die neu entwickelte ddPCR-Methode setzt sich aus zwei Nachweissystemen zusammen: das Erste zielt auf einen Sequenzunterschied zwischen Dinkel und Weizen im γ-gliadin-Gen. Da dieses Nachweissystem jedoch die gleiche Problematik wie die bestehende qPCR-Nachweismethode aufweist, wurde ein zweites Nachweissystem entwickelt, das den Austausch eines einzelnen DNA-Bausteins im sogenannten Q-Locus des Dinkels detektiert. Beide Nachweissysteme funktionieren besser bei Dinkel-DNA als bei Weizen-DNA, wodurch Dinkel-positive Reaktionen von Weizen-positiven Reaktionen und negativen Reaktionen aufgrund der Fluoreszenzstärke unterschieden werden können (siehe Abbildung). Im Rahmen der Methodenentwicklung wurden 84 Dinkelsorten, 73 Weizensorten und auch andere Getreidearten untersucht. Mit dem Nachweissystem für den Q-Locus konnten alle 84 Dinkelsorten und alle 73 Weizensorten korrekt zugeordnet werden, während 10 von 84 Dinkelsorten mit der γ-gliadin-Nachweismethode wie Weizen reagierten. Einkorn und Emmer reagieren mit der Q-Locus-Nachweismethode wie Dinkel, mit der γ-gliadin-Nachweismethode gar nicht. Die Kombination dieser beiden Nachweisverfahren ermöglicht somit nicht nur die korrekte Zuordnung von Dinkel und Weizen, sondern gibt auch einen Hinweis auf das Vorhandensein „atypischer“ Dinkelsorten, in die Weizen eingekreuzt wurde. Darüber hinaus können auch Produkte aus Einkorn und Emmer auf Weizenbeimengungen untersucht werden.
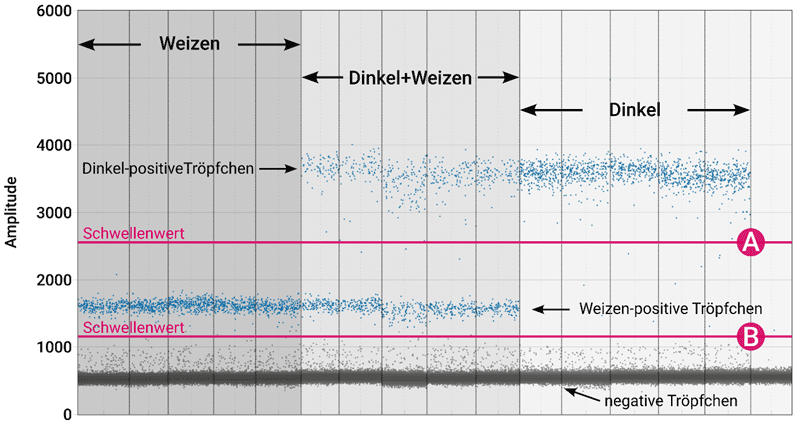
Abbildung: Exemplarische Darstellung einer Auswertung des Q-Locus-Nachweissystems in der digitalen PCR zu Bestimmung des Weizenanteils in Dinkelprodukten.
Die Punkte repräsentieren die einzelnen Tröpfchen. Ist die Ziel-DNA in einem Tröpfchen nicht vorhanden, kommt es zu keinem Fluoreszenzanstieg und die Tröpfchen werden weiter unten im Diagramm aufgetragen. Bei Weizen funktioniert das PCR-Nachweissystem schlechter als bei Dinkel, weshalb der Fluoreszenzanstieg bei vorhandener Weizen-DNA niedriger ausfällt als bei vorhandener Dinkel-DNA. Daher werden die Weizen-positiven Tröpfchen zwischen den negativen und den Dinkel-positiven Tröpfchen aufgetragen. Wird nun der Schwellenwert zwischen die Weizen-positiven und Dinkel-positiven Tröpfchen gelegt (A), kann die Konzentration der Dinkel-DNA in der Probe berechnet werden. Wird der Schwellenwert zwischen die negativen und die Weizen-positiven Tröpfchen (B) gelegt, wird die Ausgangskonzentration von Dinkel- und Weizen-DNA („Dinkel + Weizen-DNA“) berechnet. Das Verhältnis dieser beiden Konzentrationen erlaubt die Berechnung des Weizenanteils der Probe. Links sind Proben dargestellt, die nur Weizen-DNA enthalten, in der Mitte sind Proben dargestellt, die Dinkel- und Weizen-DNA enthalten und rechts sind Proben dargestellt, die nur Dinkel enthalten. Ganz rechts ist eine Negativkontrolle dargestellt.
Im Jahr 2022 fand ein nationaler Ringversuch mit verschiedenen Matrices (z. B. Mehl, Brot, Kekse, Nudeln) statt, um die entwickelte Methode zu validieren. Dieser Ringversuch, an dem auch das LGL teilgenommen hat, wurde im Rahmen der nach § 64 LFGB (Lebensmittel- und Futtermittelgesetzbuch) beim Bundesamt für Verbraucherschutz und Lebensmittelsicherheit eingerichteten Arbeitsgruppe „Molekularbiologische Methoden zur Pflanzen- und Tierartendifferenzierung durchgeführt. Die Ergebnisse dieses Ringversuchs werden für 2023 erwartet.
Untersuchung von Dinkelprodukten am LGL
Das LGL wendet die neue Methode seit 2021 zur Bestimmung des Weizenanteils in verschiedenen Dinkelprodukten an.
In den Jahren 2021 und 2022 wurden 80 Dinkelerzeugnisse (Backwaren, Teigwaren, Mehle, Dinkelkörner) untersucht. Von den untersuchten Dinkelteigwaren waren zwei Proben teilweise mit Weizenmehl vermischt, die eine zu 27 % und die andere sogar zu zwei Dritteln. Eine Probe Dinkelteigwaren und eine Probe der untersuchten Dinkelkörner waren wegen Kennzeichnungsmängeln und Werbeaussagen zu beanstanden, nicht jedoch wegen Weizenbeimischungen.
Literatur
- [1] Köppel, R., Guertler, P., & Waiblinger, H. U. (2021). Duplex droplet digital PCR (ddPCR) method for the quantification of common wheat (Triticum aestivum) in spelt (Triticum spelta). Food Control, 130, 108382
- [2] Leitsätze für Brot und Kleingebäck. Neufassung vom 01.04.2021 (BAnz AT 06.05.2021 B2, GMBl 29/2021 S. 654-659) zuletzt geändert durch die Bekanntmachung vom 13.09.2022 (BAnz AT 04.10.2022 B2, GMBl 36/2022, S. 824)
- [3] Leitsätze für Teigwaren; Neufassung vom 14.09.2022 (BAnz AT 18.10.2022 B3, GMBl 37/2022, S. 835-837)
- [4] Mayer, Franz, et al. Use of polymorphisms in the γ-gliadin gene of spelt and wheat as a tool for authenticity control. Journal of Agricultural and Food Chemistry 60.6 (2012): 1350-1357.


