Forschungsprojekt: Gleichzeitiger Nachweis mehrerer gentechnisch veränderter Pflanzen mit der 'Multiplex ligationsabhängigen Sondenamplifikation' (MLPA)
Kurzbeschreibung
Die Zahl an gentechnisch veränderten Pflanzen (GVP), die weltweit angebaut werden, hat seit Beginn der Kommerzialisierung im Jahr 1996 stetig zugenommen. Dies stellt eine große Herausforderung für die amtliche Lebensmittel-, Futtermittel- und Saatgutüberwachung dar. Der steigenden Zahl an GVP kann nur mit Nachweissystemen begegnet werden, die simultan mehrere dieser Pflanzen nachweisen können. Eine dieser Methoden stellt die 'Multiplex ligationsabhängige Sondenamplifikation' (MLPA) dar.
Die MLPA basiert auf der Bindung zweier Sonden an die Ziel-DNA. Diese Sonden bestehen aus einer Hybridisierungssequenz, über die die Bindung an die DNA erfolgt, eine Stuffer-Sequenz, welche die Länge der Sonde definiert, und einer universellen Primerbindestelle, die bei jedem Sondenpaar gleich ist. Die beiden Sonden binden nebeneinander an die Ziel-DNA und werden durch das Enzym Ligase miteinander verknüpft. Anschließend binden Fluoreszenz (FAM)-markierte Primer an die universellen Primerbindestellen der Sonden und es folgt eine PCR-Reaktion mit 40 Zyklen. Sondenpaare werden so erstellt, dass jedes Sondenpaar eine spezifische Gesamtfragmentlänge hat. Dadurch entstehen bei der Verwendung von mehreren Sondenpaaren in einer Reaktion Amplicons mit unterschiedlicher Fragmentlänge. Diese Amplicons können dann über eine Kapillarelektrophorese oder ein hochauflösendes Gel aufgetrennt werden. Der Ablauf der LPA ist in Abbildung 1 schematisch dargestellt.
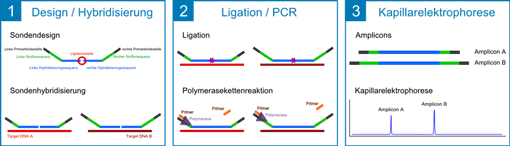
Abbildung 1: schematische Darstellung des Ablaufs einer LPA-Reaktion – 1) die linke und rechte Sonde bindet an die Ziel-DNA und wird 2) ligiert. Anschließend binden universelle Primer an die Bindestelle der Sonde und die DNA-Polymerase amplifiziert den zweiten DNA-Strang. 3) Nach 40 PCR-Zyklen entstehen Amplicons mit unterschiedlicher Länge, die über eine Kapillarelektrophorese ihrer Größe nach aufgetrennt und dadurch unterschieden werden können.
Im Rahmen dieses Projekts wurden Module für die MLPA entwickelt, anhand derer die wichtigsten gentechnisch veränderten Mais-, Raps-, Soja-, Reis- und Kartoffelevents nachgewiesen werden können. Zusätzlich wurde ein Screening-Modul entwickelt, um die häufigsten in GVP eingebrachten Genelemente simultan nachweisen zu können. Weiterhin wurde für die Aufarbeitung der DNA vor der MLPA-Analyse eine automatisierte DNA-Extraktionsmethode entwickelt und in den MLPA-Ablauf integriert.
Laufzeit: 2010 bis 2013
Finanzielle Förderung: Dieses Forschungsprojekt wird durch das Bayerische Staatsministerium für Umwelt und Verbraucherschutz (StMUV) gefördert.

