Empfindlichkeitstestung von Bakterien am Beispiel von zwei Krankheitsbildern beim Rind in Bayern
Abstract
Die Empfindlichkeitstestung von Bakterien, kurz Antibiotikaresistenztestung, nimmt in der bakteriologischen Diagnostik einen bedeutenden Stellenwert ein. Hintergrund ist die wirksame Behandlung bakterieller Infektionskrankheiten bei Mensch und Tier. Das LGL erstellt jährlich mehrere tausend Antibiogramme von Bakterien, die zuvor aus erkrankten Nutztieren isoliert wurden (Abbildung 1). Eine gesonderte statistische Auswertung von Daten aus den letzten Jahren erfolgte hier für die Tierart Rind. Eine Vielzahl der untersuchten Isolate zeigten dabei Mehrfach- oder Multiresistenzen. Weiterhin wurden Trends der Resistenzentwicklung ausgewertet und Daten einzelner Betriebsmerkmale und Haltungsformen erhoben. Die Auswertung und Veröffentlichung solcher Daten zu bakteriellen Resistenzen stellt einen wichtigen Beitrag für Maßnahmen zur Verbesserung der Tiergesundheit und damit auch der Lebensmittelsicherheit im Rahmen des One-Health-Konzepts dar.
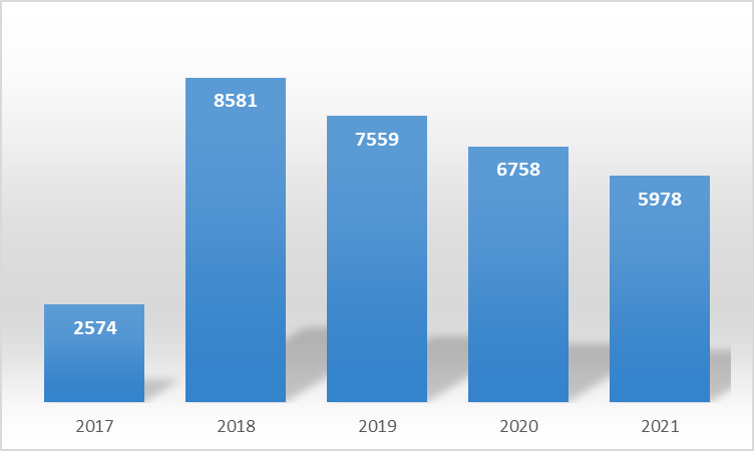
Abbildung 1: Anzahl der Empfindlichkeitstestungen von Bakterien bei Nutztieren am LGL; der Anstieg von 2017 auf 2018 ist mit der Änderung der Tierärztlichen Hausapothekenverordnung zu erklären.
Hintergrund
In deutschen Rinderbeständen sorgen insbesondere zwei Krankheitsbilder für hohe wirtschaftliche Verluste, die Jungtier-Enteritis (Kälberdurchfall) und die Enzootische Bronchopneumonie (sog. Rindergrippe). Bei beiden Erkrankungen sind neben Viren insbesondere pathogene Bakterien beteiligt. Bei schweren Krankheitsverläufen ist die Behandlung mit antibiotisch wirksamen Medikamenten unumgänglich. Um geeignete antibiotische Wirkstoffe auswählen zu können, werden die beteiligten Infektionserreger zunächst mit labordiagnostischen Standardmethoden molekular oder kulturell bestimmt. Im Anschluss erfolgt bei krankheitsursächlichen Bakterienisolaten regelmäßig die Bestimmung von Antibiotikaresistenzen (Abbildung 2). Das LGL erstellt im Jahr mehrere tausend Antibiogramme mittels Mikrobouillondilution (Abbildung 2). Aktuelle klinische Grenzwerte für die Einstufung eines Erregers als „sensibel“, „intermediär“ oder „resistent“ werden aus den international anerkannten Regelwerken des Clinical and Laboratory Standards Institute (CLSI) übernommen. In die hier beschriebene Studie zur „Antibiotikaresistenz bei Infektionserregern vom Rind“ hat das LGL knapp 10.000 Antibiogramme aus den Jahren 2017 bis 2021 einbezogen. Zudem wurden epidemiologische Daten, wie Alter und Geschlecht der Tiere, sowie Haltungsform und Tierzahl im Betrieb erhoben und statistisch ausgewertet.

Abbildung 2: Reinkultur von Pasteurella multocida (links) und optische Auswertung eines Antibiogramms (rechts).
Ergebnisse
Für das Krankheitsbild der Jungtier-Enteritis fielen nahezu alle von erkrankten Tieren eingesandten Proben mit enteropathogenen Escherichia coli (EPEC) vom Kalb aus den Jahren 2017 bis 2021 in die sogenannten Kategorien „multidrug-resistant“ (MDR, 84%) und „extensively drug-resistant“ (XDR, 16%). Die Resistenzlage gegen therapeutisch relevante Antibiotika beim Kälberdurchfall blieb im genannten Fünfjahreszeitraum zwar hoch, aber weitgehend stabil [1].
Eingesandte Proben von an Enzootischen Bronchopneumonie (EBP) erkrankten Tieren enthielten Keime aus der Gruppe der Pasteurellen, wie z. B. Pasteurella multocida, Mannheimia haemolytica und Bibersteinia trehalosi. Weiterhin konnten häufig Trueperella pyogenes und gelegentlich Mycoplasma Spezies isoliert werden. Die Pasteurella multocida Isolate zeigten innerhalb des genannten Fünfjahreszeitraumes einen signifikanten Anstieg der intermediären und resistenten Isolate für die Substanzen Tulathromycin (von 6 % auf 26 %) und Tetracyclin (von 19 % auf 57 %). Gleichzeitig stieg der Anteil mehrfachresistenter Isolate signifikant von 4 % auf 23 % an.
Die Auswertung der epidemiologischen Daten zur EBP ergab, dass der Anteil der mehrfachresistenten Isolate in Mastbetrieben mit 22 % höher war als in milchliefernden Betrieben mit 7 %. Zudem konnte gezeigt werden, dass die Mehrfachresistenz mit 27 % in größeren Betrieben mit mehr als 300 Tieren höher war, als in kleineren Betrieben, die nur 100 oder weniger Tiere hielten (10 %) (Abbildung 3) [2].
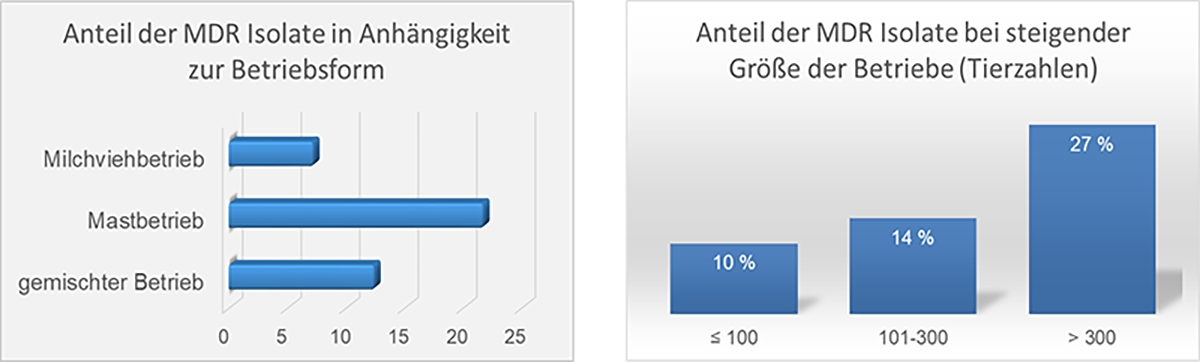
Abbildung 3: Auswertung zur Antibiotikaresistenz bei Bakterien, die im Rahmen der Enzootischen Bronchopneumonie des Rindes isoliert wurden in Bezug auf Betriebsparameter. MDR: multidrug resistance
Fazit
Bei den Empfindlichkeitstestungen werden Daten über die isolierten Bakterienspezies, die verschiedenen antimikrobiell wirksamen Substanzen, Tierart und Alter des erkrankten Tieres, das betroffene Organsystem, sowie Daten zu Betriebsform und -größe erhoben. Zwar erschwert sich mit jeder dieser Variablen eine standardisierte Übertragung in Auswertungssysteme und damit der Vergleich von Daten aus unterschiedlichen Quellen, Trotzdem stellt die in der Veterinärmedizin gesetzlich vorgeschriebene Resistenzdiagnostik einen wichtigen Baustein nicht nur für die Verbesserung der Tiergesundheit in den jeweiligen Tierhaltungen dar, sondern kann auch für Einschätzungen der allgemeinen Resistenzlage bei der jeweiligen Tierart und Erkrankung genutzt werden.
Literatur
- Feuerstein A, Scuda N, Klose C, Hoffmann A, Melchner A, Boll K, Rettinger A, Fell S, Straubinger RK, Riehm JM. Antimicrobial Resistance, Serologic and Molecular Characterization of E. coli Isolated from Calves with Severe or Fatal Enteritis in Bavaria, Germany. Antibiotics (Basel). 2021 Dec 27;11(1):23. doi: 10.3390/antibiotics11010023.
- Melchner A, van de Berg S, Scuda N, Feuerstein A, Hanczaruk M, Schumacher M, Straubinger RK, Marosevic D, Riehm JM. Antimicrobial Resistance in Isolates from Cattle with Bovine Respiratory Disease in Bavaria, Germany. Antibiotics (Basel). 2021 Dec 15;10(12):1538. doi: 10.3390/antibiotics10121538.
Mehr zu diesem Thema
Allgemeine Informationen zum Thema
Service/Aufgaben
Untersuchungsergebnisse
- Untersuchungsergebnisse 2017
- Untersuchungsergebnisse 2016
- Untersuchungsergebnisse 2015
- Untersuchungsergebnisse 2014
- Untersuchungsergebnisse 2013
- Untersuchungsergebnisse 2011
- Untersuchungsergebnisse 2010
- Untersuchungsergebnisse 2009
- Untersuchungsergebnisse 2008


