"GenoSalmSurv" – Projekt zur genombasierten Surveillance von Salmonellen mittels Next Generation Sequencing
Abstract
Das vom Bundesgesundheitsministerium (BMG) geförderte Projekt „GenoSalmSurv“ (genombasierte Surveillance von Salmonellen) wurde als 3-jährige Kooperation zwischen mehreren Bundesbehörden und dem LGL durchgeführt und endete im Oktober 2022.
Ziel des Projektes war der Aufbau einer integrierten, genombasierten Surveillance für Salmonellen, basierend auf Daten des Next Generation Sequencing (NGS), um Ausbrüche durch genetische Verwandtschaft von Isolaten sektorenübergreifend und überregional aufzuklären. Dazu gehörte die strukturierte Zusammenführung der Daten der unterschiedlichen Sektoren (Human, Lebensmittel und Veterinär), sowie der genombasierten Typisierungsdaten mit epidemiologischen Daten. Im Projekt wurden harmonisierte Arbeitsabläufe und auf lizenzfreien Softwares basierende Genomanalyseverfahren zur Clustererkennung, Erregerüberwachung und Bewertung entwickelt. Darüber hinaus wurden weitere Behörden befragt und geschult, um die genombasierte Überwachung und Ausbruchsanalysen zukünftig vermehrt zugänglich zu machen.
Hintergrund
Die Salmonellose ist in Deutschland die zweithäufigste bakterielle Magen-Darm-Infektion mit einer hohen Hospitalisierungsrate, wobei es immer wieder zu regionalen, aber auch internationalen, lebensmittelbedingten Ausbrüchen kommt. Verursacher dieser Erkrankung sind Bakterien der Gattung Salmonella.
Next Generation Sequencing (NGS) ist eine molekulare Hochdurchsatztechnologie, die mit einer hohen Auflösung komplette Genome von Krankheitserregern entschlüsseln kann. Durch den Vergleich der erzeugten Genomdaten und so detektierter Unterschiede oder Ähnlichkeiten zueinander ist die Identifizierung von Clustern und damit eine molekulare Erregerüberwachung und Infektionsquellenanalyse möglich.
Um überregional und/oder sektorübergreifend (zwischen Patient, Lebensmittel und/oder Nutztier) Cluster zu identifizieren sind standardisierte Auswerteverfahren und ein gemeinsamer Abgleich der Erregergenomdaten über alle betroffenen Sektoren hinweg nötig.
Das vom Bundesministerium für Gesundheit geförderte 3-jährige Projekt zur genombasierten Surveillance von Salmonellen GenoSalmSurv zielte darauf ab, ein funktionierendes Modell für die integrierte, molekulare, sektorübergreifende sowie überregionale Überwachung von Salmonella spp. zu etablieren. Projektpartner waren die Bundesbehörden des Gesundheits-, des Lebensmittels- und des Veterinärbereichs (Robert Koch Institut (RKI), Bundesinstitut für Risikobewertung (BfR), Bundesamt für Verbraucherschutz und Lebensmittelsicherheit (BVL), Friedrich-Löffler-Institut (FLI)) und als Vorreiter auf Bundeslandebene das LGL.
Ergebnisse
Zu Beginn des Projektes fand eine deutschlandweite Befragung von weiteren Landes- und Bundesämtern bezüglich des Status quo und der Bedürfnisse für molekulare Surveillance statt (3).
Basierend auf den Ergebnissen erarbeiteten die Projektpartner harmonisierte Laborverfahren für die Erregersequenzierung per NGS und implementierten bioinformatische Datenauswerte-Pipelines, die frei verfügbare, sogenannte Open-Source-Softwares nutzen. Kernstück dabei war die cgMLST-Analyse (core genome Multi Locus Sequence Typing), die jedes sequenzierte Genom mittels eines definierten Sets von mehreren Tausend erregerspezifischen Kerngenomabschnitten typisiert und daraus Allelprofile erstellt. Die so erzielten standardisierten Allelprofile benötigen im Gegensatz zu den kompletten Genomdaten wenig Speicherplatz und können einfach ausgetauscht werden.
Die Projektpartner etablierten gemeinsame Routinepraktiken für den Austausch genomischer und epidemiologischer Daten und für eine einheitliche Interpretation der Ergebnisse (4). Dies beinhaltete auch abgestufte Grenzwerte für Allel-Distanzen der cgMLST-Typisierungsergebnisse, um die sequenzierten Salmonella-Genome Ausbruchsclustern zuzuordnen oder Zusammenhänge auszuschließen. Des Weiteren implementierten die Partner ein automatisiertes Namensschema für die identifizierten Cluster mit ihren zugehörigen Abstandsgrenzen (Vornamen plus griechische Buchstaben), um die Kommunikation zwischen den einzelnen Projektpartnern zu erleichtern.
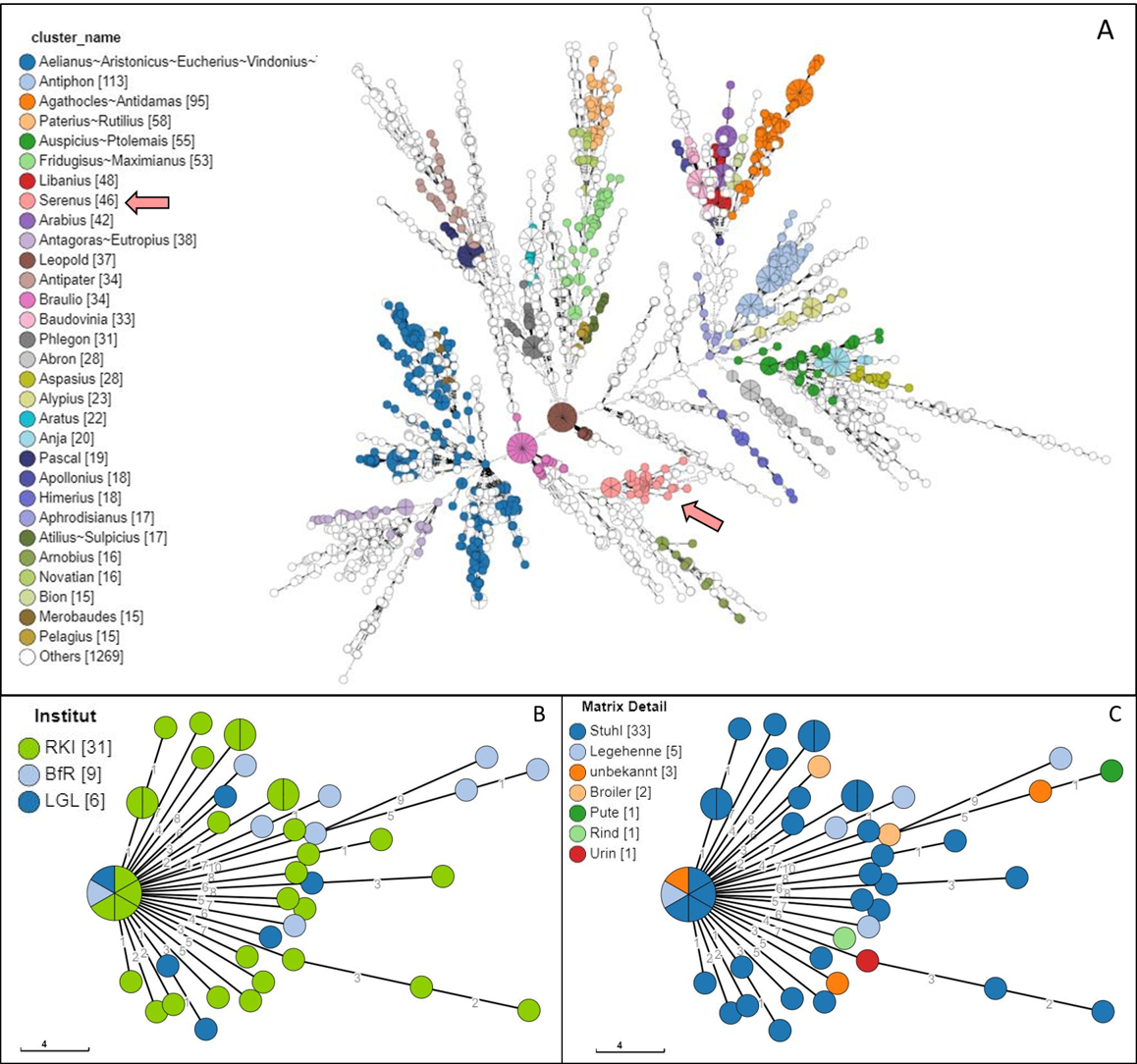
Das Projekt umfasste zwei Echtzeit-Sequenzierphasen, in der die Projektpartner die im Gesundheitsbereich relevantesten Salmonella-Serotypen (unter anderem S. Typhimurium, S. Enteritidis, S. Infantis) aus verschiedenen Quellen (Mensch, Tier, Lebensmittel, Futtermittel, Umwelt) regelmäßig sequenzierten. Das LGL wie auch die anderen Projektpartner führten Sequenzierung und bioinformatische Analysen der Sequenzdaten der eigenen Isolate dezentral, mittels der im Projekt entwickelten Software-Pipelines (AQUAMIS (1), BakCharak (4) und chewieSnake (2)) durch. Die Ergebnisse der cgMLST-Typisierungsanalyse wurden dann standardisiert in einer gemeinsamen Datenbank ausgetauscht, grafisch in phylogenetischen Bäumen visualisiert (Abbildung) und bezüglich identifizierter Cluster und sich daraus ergebender Handlungsmaßnahmen bewertet. Bei der Bewertung sind dabei neben den Genomdaten immer auch die epidemiologischen Zusammenhänge zu berücksichtigen.
Außerdem organisierte das LGL zusammen mit den Projektpartnern im Mai 2022 einen Bioinformatik-Workshop für internes und externes Fachpublikum. Ziel war es, Mitarbeitenden der Untersuchungseinrichtungen der Länder theoretischen und praktischen Einblick in die Auswertung und Interpretation von Ganzgenomsequenzdaten von Salmonellen unter Verwendung der entwickelten Pipelines zu geben. Zusätzlich wurden Online Schulungsvideos (Screencasts) der einzelnen Analyseschritte erstellt, die auf der LGL- und BfR-Internetseite veröffentlicht sind.
Die Abbildung zeigt Beispiele von phylogenetischen Bäumen („Minimum Spanning Tree“; MST) zur Clusteranalyse. Jedes Salmonella-Genom wird im MST als Kreis dargestellt, sowie die dazwischenliegenden genetischen Allel-Distanzen auf den Verbindungslinien. Genetisch identische Genome werden als „Tortenstücke“ eines Kreises dargestellt. (A) Übersichtsbaum einer Clusteranalyse, eingefärbt nach ausgewählten Clusternamen. Als Beispiel ist Cluster „Serenus“ (in (A) roséfarben und mittels Pfeil markiert) in (B) und (C) vergrößert dargestellt und die enthaltenen Genome entweder (B) nach dem Partnerinstitut, bei dem sie sequenziert wurden oder (C) der Probenmatrix eingefärbt. Aus (B) und (C) wird deutlich, dass es sich bei dem Cluster „Serenus“ um ein gemischtes Cluster handelt, dessen Isolate aus verschiedenen Quellen und von verschiedenen Partnerinstituten stammen.
Fazit und Maßnahmen
Das entwickelte Vorgehen ermöglicht, Isolate schnell und effizient bei einem Projektpartner zu sequenzieren, durch die Zusammenarbeit zwischen den verschiedenen Projektpartnern mit bundesweit sequenzierten Isolaten abzugleichen und so Zusammenhänge rasch zu erkennen.
Next Generation Sequencing ist dabei das optimale Tool für genombasierte Salmonellen-Surveillance und Ausbruchsanalytik und verfügt über die höchstmögliche analytische Auflösung. Eine gemeinsame Datenbank sowie standardisierte und harmonisierte Abläufe, von der Datengenerierung bis hin zur Clusteridentifizierung und Bewertung, sind – immer unter Einbeziehung der Epidemiologie – wichtig für die überregionale und sektorübergreifende genomische Surveillance.
Das LGL wird auch nach Projektende die genombasierte Salmonellen-Surveillance und die gemeinsame GenoSalmSurv-Datenbank weiter nutzen. Es besteht der Wunsch die Echtzeitsequenzierung und die genombasierte Salmonellen-Surveillance auf weitere Salmonella-Serovare auszuweiten und die GenoSalmSurv-Datenbank auch für weitere Partner (z.B. Landesinstitute anderer Bundesländer) zugänglich zu machen. Auch für die Ausweitung auf weitere Krankheitserreger gibt es mittlerweile ähnliche Projekte und Kooperationen.
Bei der Identifikation von relevanten Salmonellenclustern wird von den sequenzierenden Partnern eine Meldung der Fälle sowie des jeweils zugehörigen Clusters an weitere Landes- und Bundesstellen für Epidemiologie und Vollzug durchgeführt. So können bei Bedarf weitere Schritte, wie die Befragungen von Erkrankten zur Bestätigung von potenziellen Zusammenhängen oder ursächlichen Lebensmitteln, oder aber auch Vollzugsmaßnahmen wie Betriebskontrollen oder weitere Probengewinnung, veranlasst werden.
Literaturverzeichnis
- Deneke C., Brendebach H., Uelze L., Borowiak M., Malorny B., Tausch S.H.; 2021. Genes 2021, 12(5), 644. Species-Specific Quality Control, Assembly and Contamination Detection in Microbial Isolate Sequences with AQUAMIS; https://doi.org/10.3390/genes12050644
- Deneke C., Uelze L., Brendebach H., Tausch S.H., Malorny B.; Front. Microbiol.; 2021. Front. Microbiol., 28 May 2021. ‘Decentralized Investigation of Bacterial Outbreaks Based on Hashed cgMLST’; https://doi.org/10.3389/fmicb.2021.649517
- Pietsch M., Simon S., Richter A., Malorny B., Uelze L., Hepner S., Dangel A., Sing A., Huber I., Busch U., Linde J., Methner U., Becker N., Werner G., Mellmann A., Fruth A., Flieger A.; 2023. Bundesgesundheitsblatt - Gesundheitsforschung - Gesundheitsschutz, 66: 75-83. Bestandsaufnahme der verfügbaren und aktuell eingesetzten Typisierungsmethoden einschließlich genombasierter Verfahren von Zoonoseerregern am Beispiel von Salmonella enterica [Assessment of available and currently applied typing methods including genome-based methods for zoonotic pathogens with a focus on Salmonella enterica]'; https://doi.org/10.1007/s00103-022-03622-y
- Uelze, L., N. Becker, M. Borowiak, Busch U., Dangel A., Deneke C., Fischer J., Flieger A., Hepner S., Huber I., Methner U., Linde J., Pietsch M., Simon S., Sing A., Tausch S.H., Szabo I., Malorny B.; 2021. Front. Microbiol., 10 February 2021. 'Toward an Integrated Genome-Based Surveillance of Salmonella enterica in Germany'; https://doi.org/10.3389/fmicb.2021.626941