- Startseite >>
- Gesundheit >>
- Infektionsschutz >>
- Bayvoc
Erweiterung der Bayerischen Plattform zur Überwachung von SARS-CoV-2-Varianten BayVOC
Abstract
SARS-CoV-2 zirkuliert weiter weltweit, sodass immer wieder neue Varianten auftreten können. Nur durch eine engmaschige virologische Überwachung (= Surveillance) lassen sich Varianten mit hohem Ausbreitungs- und Gefährdungspotenzial rasch erkennen. Der Freistaat Bayern unterstützt den Ausbau einer breiten virologischen Surveillance durch die Förderung von drei Projekten: (1) Das Verbundprojekt „Bay-VOC“ zur Überwachung von SARS-CoV-2-Virusvarianten wird gestärkt und eng mit (2) BIS+C, dem Bayern Influenza/Corona-Sentinel von bayerischen Arztpraxen und (3) dem Abwassermonitoring auf SARS-CoV-2 verzahnt. Ziel ist es, die Projekte zu einem effizienten Infektionsfrühwarnsystem zu bündeln, das einen zentralen Beitrag zur vorausschauenden Risikobewertung zum Schutz der bayerischen Bevölkerung leisten soll.
Hintergrund und Entwicklung
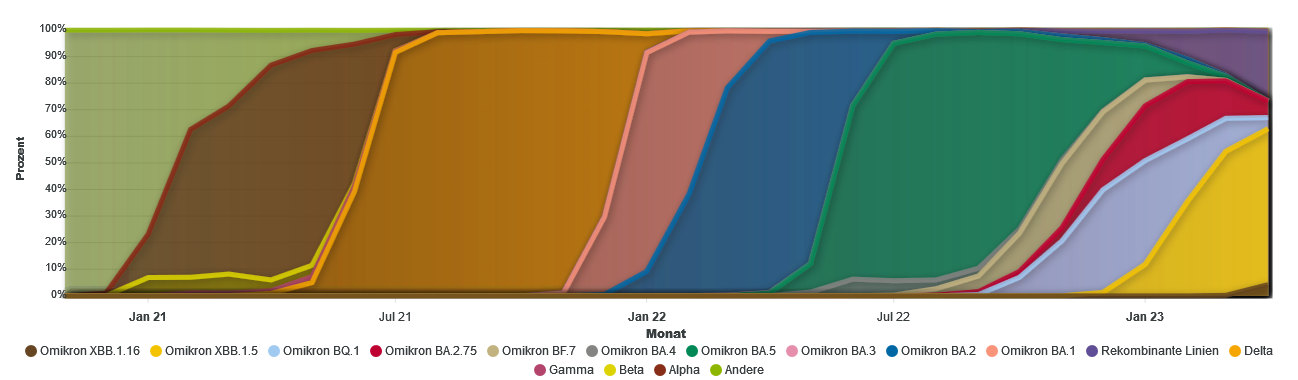
Seit seinem erstmaligen Nachweis Ende 2019 hat sich SARS-CoV-2 weltweit rasch ausgebreitet und mehrere Millionen Menschen infiziert. Die Dynamik der Pandemie sowie das Auftauchen neuer „besorgniserregender Varianten“ (VOC: Variants of Concern) wie der Alpha-, Delta- und Omikron-Varianten, die z.B. mit erhöhter Übertragbarkeit und teils auch erhöhter Krankheitsschwere einhergehen, haben gezeigt, wie wichtig eine zeitnahe Analyse der zirkulierenden Virusvarianten ist. Nur durch die Sequenzierung des SARS-CoV-2-Virusgenoms von möglichst vielen infizierten Personen ist es möglich, neu auftretende Varianten des Virus zuverlässig und schnell zu erkennen. Daher werden weltweit zunehmend Next Generation Sequencing (NGS)-Verfahren zur Analyse von SARS-CoV-2-Genomen verwendet. Gleichzeitig kann so auch ein besserer Überblick über die zirkulierenden Virusvarianten und deren Verbreitungsmuster gewonnen werden. Vor diesem Hintergrund hat das Bundesministerium für Gesundheit am 19. Januar 2021 die Coronavirus-Surveillanceverordnung erlassen. Damit wurden Anreize aber auch Richtlinien für den Ausbau der SARS-CoV-2-Genomsequenzierung festgelegt und so die Grundlage für eine bundesweite systematische und kontinuierliche Überwachung der SARS-CoV-2-Virusentwicklung in Deutschland geschaffen. Ergänzend dazu hat die Bayerische Staatsregierung zum Ausbau der molekularen Surveillance von SARS-CoV-2 in Bayern am 2. Februar 2021 das landesgeförderte Verbundprojekt Bay-VOC beschlossen. Die an Bay-VOC beteiligten Projektpartner setzen sich aus dem öffentlichen Gesundheitsdienst, vertreten durch das LGL und den sechs bayerischen Universitätsklinika zusammen und bündeln somit die virologisch-infektionsepidemiologische Expertise des Freistaats. Die erhobenen bayerischen SARS-CoV-2-Sequenzdaten werden nicht nur in einer gemeinsamen digitalen bayerischen VOC-Datenbank gesammelt, sondern auch an das Robert-Koch Institut (RKI) übermittelt, wo die Daten für die nationale Surveillance zur Verfügung stehen. Die engmaschige Überwachung durch das Bay-VOC-Projekt ermöglichte so die frühzeitige und zuverlässige Erkennung des Auftretens der Delta-Variante im April 2021 und der Omikron-Variante im November 2021 in Bayern (Abbildung 1).
Abbildung 1. Prozentualer Anteil besorgniserregender SARS-CoV-2-Varianten in Bayern.
Daten zur Häufigkeit von SARS-CoV-2-Varianten, die in Bayern im Zeitraum von November 2020 bis April 2023 durch Ganzgenomsequenzierung aus Patientenabstrichen ermittelt wurden. Die Daten stammen von den bayerischen Universitätskliniken und dem Landesamt für Gesundheit und Lebensmittelsicherheit (LGL). Zusätzlich sind Sequenzdaten der bayerischen Labore enthalten, die vom Robert Koch-Institut (Robert Koch-Institut (2023)) veröffentlicht werden. Der zunächst vorherrschende SARS-CoV-2 Wildtyp wurde Anfang 2021 durch die Alpha-Variante ersetzt. Von Juni bis Dezember 2021 war die Delta-Variante dominant. Seit November 2021 wird das Infektionsgeschehen von verschiedenen Omikron Subvarianten beherrscht: Zuerst von November 2021 bis Februar 2022 von BA.1. Folgend dominierte bis Mai 2022 BA.2 und wurde von BA.5 abgelöst. Bis heute (Stand: April 2023) dominieren die Sublinien BA.2 und BA.5, den größten Anteil macht die rekombinante BA.2 Sublinie XBB.1.5 aus. Die Daten geben das Datum (KW oder Monat) der Probenahme an.
Ergebnisse
Bis heute sind in der Bay-VOC-Datenbank über 25.000 SARS-CoV-2-Ganzgenomsequenzen aus Abstrichen von Patientinnen und Patienten erfasst, über 12.000 davon wurden alleine von der NGS Core Unit am LGL bearbeitet. Neben den Daten der Projektpartner fließen auch die vom RKI zur Verfügung gestellten SARS-CoV-2-Sequenzdaten aller bayerischen Labore in die Bay-VOC-Datenbank ein. So kann die Verteilung der in Bayern vorkommenden SARS-CoV-2-Varianten bestmöglich und zeitnah erfasst werden. Zur Information der Öffentlichkeit betreibt das Bay-VOC-Projekt eine Internetseite, die über die neuesten SARS-CoV-2-Varianten informiert und die Variantenverteilung in Bayern darstellt. Das zunächst auf einen Zeithorizont von zwei Jahren ausgelegte Bay-VOC-Projekt wurde 2022 weiter bis Ende 2024 verlängert und ausgebaut. So soll die bereits aufgebaute Wissens- und Laborinfrastruktur weiter genutzt und um zusätzliche Bausteine der virologischen Surveillance in Bayern erweitert werden. Zu diesem Zweck wird das Bay-VOC-Projekt mit dem Bayerischen Influenza-/Coronavirus Sentinel (BIS+C) verknüpft und ein Abwassermonitoring auf SARS-CoV-2 in Bayern aufgebaut und in Bay-VOC integriert.
Bayerisches Influenza-Sentinel BIS+C
Zur Überwachung viraler Atemwegserreger in Bayern erhebt das LGL bereits seit 2009 im Rahmen des Bayerischen Influenza-Sentinels (BIS) während der Erkältungssaison Daten zum Auftreten und zur Verbreitung von Influenza und Respiratorischen Synzytialviren (RSV). Mit Beginn der SARS-CoV-2-Pandemie wurde das Erregerspektrum um SARS-CoV-2 erweitert (BIS+C). Zu diesem Zweck senden ausgewählte Arztpraxen wöchentlich Abstriche von Patienten mit Symptomen einer Atemwegsinfektion zur epidemiologischen und diagnostischen Analyse an das LGL. Durch die Projektförderung ist es nun möglich, eine größere Anzahl an Abstrichen ganzjährig zu untersuchen. Zudem konnte die Zahl der teilnehmenden Praxen erhöht werden, was einen besseren Überblick über das Infektionsgeschehen ermöglichen soll. In die Bay-VOC-Datenbank fließen auch die im Rahmen des BIS+C-Projekts gewonnenen SARS-CoV-2-Sequenzdaten mit ein, was die bayernweite Analyse der SARS-CoV-2-Varianten weiter ergänzt.
Abwassermonitoring
Das Abwassermonitoring ist ein weiterer Baustein der virologischen Surveillance in Bayern. Hierzu werden die kommunalen Abwässer verschiedener Städte und Gemeinden auf das Vorhandensein von SARS-CoV-2 untersucht, welches unter anderem über den Stuhl infizierter Personen ins Abwasser gelangt und mittels molekularbiologischer Methoden nachgewiesen werden kann. Während mithilfe von PCR-Tests die Virusmenge im Abwasser bestimmt werden kann, erlaubt die NGS-basierte Sequenzierung den Nachweis von SARS-CoV-2-Genfragmenten im Abwasser, die wiederum Hinweise auf das Vorhandensein verschiedener SARS-CoV-2-Varianten im Abwasser geben können. Anhand dieser Daten können Rückschlüsse gezogen werden, ob und falls ja, welche Corona-Infektionen in der Bevölkerung eines bestimmten Gebiets zu- oder abnehmen und dies unabhängig von der aktuell gültigen SARS-CoV-2-Teststrategie. Mithilfe von Fördermitteln des Bundes und des Freistaates Bayern ist aktuell eine flächendeckende Beprobung des Abwassers an 24 Standorten in Bayern möglich und wird derzeit für weitere Standorte vorbereitet. Das LGL ist in diesem Projekt mit der Gesamtkoordination des Abwassermonitorings betraut und führt einen Teil der Routinediagnostik durch. Über ein neues Online-Dashboard auf der Bay-VOC-Internetseite kann sich auch die Öffentlichkeit über das Vorkommen von SARS-CoV-2 im Abwasser einzelner Standorte informieren.
Fazit
Das LGL trägt als Projektpartner im Bay-VOC-Netzwerk aktiv dazu bei, die Ausbreitung von SARS-CoV-2-Varianten in Bayern zu überwachen, aber auch frühzeitig neue Coronavirus-Infektionswellen und neue besorgniserregende SARS-CoV-2-Varianten zu erkennen. Durch die Erweiterung des Bay-VOC-Projektes konnte in Bayern ein breit gefächertes Monitoring des Infektionsgeschehens und eine starke virologische Surveillance etabliert werden, welche potenziell auch auf weitere Krankheitserreger ausgedehnt werden kann.
Literatur
- Robert Koch-Institut. (2023). SARS-CoV-2 Sequenzdaten aus Deutschland (2023-01-31) [Data set]. Zenodo. https://doi.org/10.5281/zenodo.7587507 [2]
Wissenschaftliche Publikationen
- Flechsler, J., Eberle, U., Dangel, A., Hepner, S., Wimmer, C., Lutmayr, J., ... & Sing,A. (2022). Molecular SARS-CoV-2 surveillance in Bavaria shows no Omicron transmission before the end of November 2021. Infection, 50(3), 761-766.
- Jungnick, S., Hobmaier, B., Mautner, L., Hoyos, M., Haase, M., Baiker, A., ... & Fingerle, V. (2021). Detection of the new SARS-CoV-2 variants of concern B. 1.1. 7 and B. 1.351 in five SARS-CoV-2 rapid antigen tests (RATs), Germany, March 2021. Eurosurveillance, 26(16), 2100413.
- Wollschläger, P., Todt, D., Gerlitz, N., Pfaender, S., Bollinger, T., Sing, A., ... & Steinmann, J. (2021). SARS-CoV-2 N gene dropout and N gene Ct value shift as indicator for the presence of B. 1.1. 7 lineage in a commercial multiplex PCR assay. Clinical Microbiology and Infection, 27(9), 1353-e1-1353.e5.
- Heinzinger, S., Eberle, U., Angermeier, H., Flechsler, J., Konrad, R., Dangel, A., ... & Sing, A. (2022). Reciprocal circulation pattern of SARS-CoV-2 and influenza viruses during the influenza seasons 2019/2020 and 2020/2021 in the Bavarian Influenza Sentinel (Germany)–CORRIGENDUM. Epidemiology & Infection, 150.
- Jungnick, S., Hobmaier, B., Paravinja, N., Mautner, L., Hoyos, M., Konrad, R., ... & Fingerle, V. (2023). Analysis of seven SARS-CoV-2 rapid antigen tests in detecting omicron (B. 1.1. 529) versus delta (B. 1.617. 2) using cell culture supernatants and clinical specimens. Infection, 51(1), 239-245.
- Jungnick, S., Hobmaier, B., Mautner, L., Hoyos, M., Haase, M., Baiker, A., ... & Fingerle, V. (2021). In vitro rapid antigen test performance with the SARS-CoV-2 variants of concern B. 1.1. 7 (Alpha), B. 1.351 (Beta), P. 1 (Gamma), and B. 1.617. 2 (Delta). Microorganisms, 9(9), 1967.
- Streibl, B. I., Lahne, H., Grahl, A., Agsten, P., Bichler, M., Büchl, C., ... & Fingerle, V. (2022). Epidemiological and Serological Analysis of a SARS-CoV-2 Outbreak in a Nursing Home: Impact of SARS-CoV-2 Vaccination and Enhanced Neutralizing Immunity Following Breakthrough Infection. Microorganisms, 10(9), 1809.