- Startseite >>
- Lebensmittel >>
- Warengruppen >>
- Wc 27 Pilze
Identifizierung von Großpilzen mit Hilfe der MALDI-TOF MS
Die Methode der MALDI-TOF MS
MALDI-TOF MS ist die Abkürzung für die matrixunterstützte Laser-Desorptions-Ionisation Flugzeit Massenspektrometrie (englisch: Matrix-assisted laser desorption/ionization time-of-flight mass spectrometry).
Mit MALDI-TOF MS können Arten anhand ihrer molekularen Fingerabdrücke, sogenannten Proteinfingerprints, identifiziert werden (Abbildung 1). Hierzu werden die Masse/Ladungsverhältnisse von Proteinen im Bereich zwischen 2.000 und 20.000 Da anhand von deren Flugzeit bestimmt. Der generierte Fingerprint wird zur Identifizierung anschließend mit in einer Datenbank hinterlegten Referenzspektren abgeglichen. Ein Ergebnis liegt innerhalb weniger Minuten vor. Die Aussagekraft und Genauigkeit der MALDI-TOF MS-Analyse hängen damit wesentlich von der Qualität und Vollständigkeit der Referenzdatenbank ab [1].
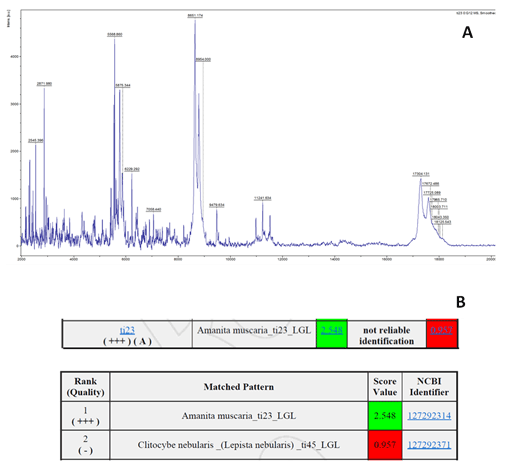
Abbildung 1: Identifizierung von Großpilzen mit dem MALDI Biotyper anhand eines Amanita muscaria -Exemplars. (A) Zunächst wird ein Masse-/Ladungsspektrum des Zielorganismus aufgenommen. Der automatische MALDI Biotyper Algorithmus vergleicht ausgewählte Peaks des Spektrums mit den Datenbankeinträgen. (B) Eine Ergebnisliste, in der die Datenbankeinträge mit den höchsten Übereinstimmungen mit abnehmender Wahrscheinlichkeit ((log)scorevalue) aufgeführt sind, wird erstellt. Zusätzlich gibt die Konsistenzkategorie (hier A, entspricht Artidentifizierung) Aufschluss über die Identifizierungsebene.
MALDI-TOF MS zur Identifizierung von Großpilzen
MALDI-TOF MS hat sich in den letzten Jahren vor allem in der Humanbakteriologie als geeignete Methode zur Identifizierung von Mikroorganismen etabliert. Mittlerweile wurde die Eignung von MALDI-TOF MS zur Identifizierung und Differenzierung von veterinärmedizinisch- und lebensmittel-relevanten Bakterien und filamentösen Pilzen durch eine Vielzahl von Studien belegt [1-6]. Am LGL wird seit Ende 2007 mit MALDI-TOF MS zur Identifizierung von Mikroorganismen gearbeitet. Die Geschwindigkeit der MALDI-TOF MS Technologie, kombiniert mit deren einfacher Handhabung, lässt den Einsatz von MALDI-TOF MS auch im Bereich der Identitätsprüfung von Lebensmitteln, wie Fischen, Fleisch oder eben Speisepilzen, attraktiv erscheinen.
Bei Speisepilzen spielt dabei nicht nur die falsche Deklaration von günstigen als hochwertige Speisepilze eine Rolle, sondern auch die Verwechslungsgefahr mit morphologisch ähnlichen Giftpilzen. Die klassische morphologische Bestimmung ist nicht nur zeitaufwändig, sondern erfordert auch mykologische Expertenkenntnisse. PCR-basierte Alternativverfahren sind auf ein enges, zuvor festgelegtes Spektrum an Arten begrenzt. MALDI-TOF MS kann hingegen auch bei kleinsten Mengen Material (ca. 15 mg) zur Artidentifizierung eingesetzt werden.
Im Rahmen des Projektes wurde die Probenaufbereitung für rohe und getrocknete Pilze optimiert und standardisiert. Anschließend wurde eine Datenbank mit Referenzspektren einer Auswahl von Speise- und Giftpilzen generiert und die Reproduzierbarkeit der Identifizierungsergebnisse überprüft. Aktuell enthält die LGL in-house Datenbank Referenzspektren von 35 Großpilzarten, darunter auch Speisepilze, Giftpilze und Heilpilze. Eine Erweiterung ist in Zusammenarbeit mit anderen Behörden geplant. Als Referenzmethoden zur Verifizierung der Identität der für die Datenbank verwendeten Großpilze wurden neben der Sequenzierung der ITS-Region makroskopische Merkmale herangezogen.
Grundsätzlich konnte gezeigt werden, dass MALDI-TOF MS geeignet ist, rohe Großpilze eindeutig und reproduzierbar zu identifizieren. Die Möglichkeit Giftpilze selbst in kleinen Mengen von Putzresten (15 mg) innerhalb von Minuten zu bestimmen, macht diese Methode nicht nur für die Lebensmittelüberwachung, sondern darüber hinaus auch zur Pilzidentifizierung beim Verdacht einer Pilzvergiftung interessant.
Ein wissenschaftlicher Beitrag zu diesem Thema wurde vom LGL auf dem 45. Deutschen Lebensmittelchemikertag (12.9-14.9.2016) in Freising präsentiert. Die Arbeit wurde dort mit einem Posterpreis ausgezeichnet [7].
Literatur
[1] Pavlovic, M., Spielmann, G., Busch, U. Huber, I. (2016) MALDI-TOF MS in der Lebensmittelmikrobiologie - Erfahrungen und Herausforderungen mit dem MALDI Biotyper, FOOD-Lab 01/2016:18–21.
[2] Pavlovic, M., Wudy, C., Zeller-Péronnet, V., Zimmermann, P., Straubinger, A., Märtlbauer, E., Busch, U., Huber, I. 2015. Identification of bacteria isolated from veterinary clinical specimens using MALDI-TOF MS. Berliner und Münchener Tierärztliche Wochenschrift 128:10–16.
[3] Pavlovic, M., Mewes, A., Maggipinto, M., Schmidt, W., Messelhäußer, U., Balsliemke, J., Hörmansdorfer, S., Busch, U., Huber, I. (2014): MALDI-TOF MS based identification of food-borne yeast isolates. Journal of Microbiological Methods 106:123–128.
[4] Pavlovic, M., Huber, I., Konrad, R., Busch, U. (2013): Application of MALDI-TOF MS for the Identification of Food Borne Bacteria. Open Microbiology Journal 7: 135–141.
[5] Hilmes, J., Pavlovic, M., Grünwald, K., Maggipinto, M., Schumacher, A., Becker, E., Kirchhoff, E., Busch, U., Huber, I. (2012): MALDI-TOF-MS in der Routineanalytik Deutsche Lebensmittelrundschau. Deutsche Lebensmittelrundschau 108:112–121.
[6] Pavlovic, M.; Konrad, R.; Huber, I.; Busch, U. (2011): Einsatz von MALDI-TOF MS zur Identifizierung von Mikroorganismen in der Lebensmittelanalytik. Deutsche Lebensmittelrundschau, Deutsche Lebensmittelrundschau 107 (3/Spezial): 9–16.
[7] Pavlovic, M., Maggipinto, M., Busch, U. (2016) Einsatz von MALDI-TOF MS zur Identifizierung von Speise- und Giftpilzen. Poster, 45. Deutscher Lebensmittelchemikertag, 12.14. September, Freising.

