Geschichte der Gentechnik und Gentechnik-Gesetzgebung
- Anwendungen der Gentechnik
- Die DNA als Trägerin der Erbinformationen
- Die Werkzeuge der Gentechnik: E. coli, Plasmide, Ligasen und Restriktionsenzyme
- Erste rekombinante DNA und erster gentechnisch veränderter Organismus
- Von der ersten Asilomar Konferenz zum Gentechnikgesetz in Deutschland
- Mehr zu diesem Thema
Anwendungen der Gentechnik
Die Anwendungen der Gentechnik nehmen weltweit immer mehr zu. Einem inoffiziellen Einteilungsschema zufolge können u. a. die Bereiche Rote, Weiße und Grüne Gentechnik unterschieden werden. Die "Rote Gentechnik" bezieht sich vor allem auf gentechnische Anwendungen in der Medizin und Pharmazie. Sie beinhaltet die Erforschung von Krankheitsursachen, die Entwicklung neuer diagnostischer und therapeutischer Verfahren sowie die Herstellung neuer Medikamente und Impfstoffe. Die "Weiße Gentechnik" beschäftigt sich mit der Optimierung industrieller Verfahren durch die Nutzung gentechnisch veränderter Mikroorganismen zum Beispiel bei der Herstellung von Enzymen und (Fein-) Chemikalien, in der Fermentation oder in der Umwelttechnik. Von "Grüner Gentechnik" spricht man bei der Anwendung gentechnischer Verfahren in der Landwirtschaft im Rahmen der Herstellung von Nutzpflanzen mit veränderten Anbaueigenschaften oder Inhaltsstoffen (Abbildung 1).
Die anwendungsbezogene Unterteilung in Rote, Weiße und Grüne Gentechnologie hat sich in weiten Teilen der Medienlandschaft durchgesetzt. Sie wird aber in keiner europäischen, nationalen oder bayerischen Rechtsvorschrift verwendet oder definiert. Gentechnikrechtliche Vorschriften unterscheiden vor allem die Anwendung genetisch veränderter Mikroorganismen in geschlossenen Systemen (EU-Richtlinie 2009/41/EG) und die absichtliche Freisetzung von genetisch veränderten Organismen in die Umwelt (EU-Richtlinie 2001/18/EG).
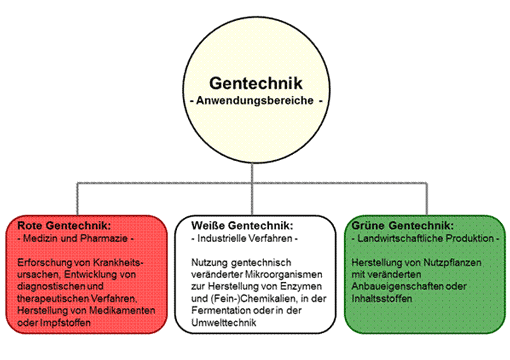
Abbildung 1: Anwendungsbereiche der Gentechnik. Die Rote Gentechnik beinhaltet die Anwendung der Gentechnik in den Bereichen Medizin und Pharmazie. Unter Weißer Gentechnik versteht man die Optimierung industrieller Verfahren unter Einsatz gentechnisch veränderter Mikroorganismen. Der Begriff Grüne Gentechnik bezieht sich auf die Anwendung gentechnischer Verfahren in der Landwirtschaft.
Die DNA als Trägerin der Erbinformationen
Im Jahre 1869 entdeckte Friedrich Miescher eine bis dato unbekannte Substanz in einem Extrakt aus Eiterzellen. Da sich diese phosphorhaltige und saure Substanz vor allem in den Zellkernen der Eiterzellen befand, nannte er sie kurz "Nuclein" (von lateinisch nucleus, der Kern). In den nachfolgenden Jahren konnte gezeigt werden, dass Mieschers "Nuclein" aus Desoxyribonukleinsäure (DNS) - oder auf Englisch: deoxyribonucleic acid (DNA) - besteht. Die Einzelbausteine der DNA sind dabei die so genannten Nukleotide, die sich jeweils aus einem Zucker (Desoxyribose), einer Phosphatgruppe und einer Base (Adenin, Guanin, Cytosin oder Thymin) zusammensetzen. Gemäß der verwendeten Base werden die Nukleotide mit A, G, C oder T abgekürzt.
Lange Zeit wurde der DNA keine nennenswerte biologische Bedeutung beigemessen und die strukturell komplexeren Proteine als wahrscheinliche Trägerinnen der Erbinformationen favorisiert. Erst 75 Jahre nach ihrer Entdeckung konnte Oswald Avery 1944 mit einem bahnbrechenden Experiment beweisen, dass die DNA die Trägerin der Erbanlagen ist (Abbildung 2).
Im Jahre 1953 postulierten James Watson und Francis Crick ihr Doppelhelixmodell der DNA-Struktur, bei dem sich zwei DNA-Einzelstränge helixartig umeinander winden. Als Grundlage ihrer Modellentwicklung dienten von Rosalind Franklin und Maurice Wilkins aufgenommene Röntgenbeugungsbilder der DNA.
Es war auch Francis Crick, der 1958 das "Zentrale Dogma der Molekularbiologie" als Hypothese verfasste, dem zufolge der Weg der genetischen Informationsweitergabe von der DNA über die RNA zum Protein verläuft.
Der genetische Code, die Regel, nach der immer drei aufeinander folgende Nukleotide der DNAbzw.RNA für eine bestimmte Aminosäure eines Proteins kodieren, wurde 1966 vollständig entschlüsselt. Er gilt dabei universell; das heißt, er ist bis auf wenige Ausnahmen bei allen Organismen identisch.
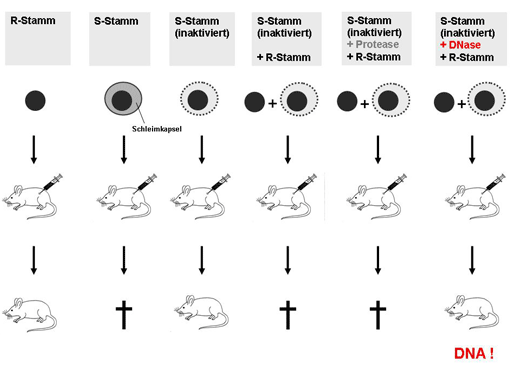
Abbildung 2: Das Pneumokokken-Experiment von Oswald Avery im Jahre 1944. Das Experiment von Avery basierte auf zwei unterschiedlichen Bakterienstämmen von Pneumokokken; einem virulenten, Schleimkapsel bildenden, so genannten S-Stamm und einem avirulenten, nicht Schleimkapsel bildenden R-Stamm. Es war bekannt, dass die Injektion von Mäusen mit dem virulenten S-Stamm in einer tödlichen Lungenentzündung resultierte. Im Gegensatz dazu überlebten die Mäuse sowohl die Injektion avirulenter R-Stamm Pneumokokken, als auch die Injektion von Zellextrakten inaktivierter S-Stamm Pneumokokken. Es war weiterhin bekannt, dass in den Zellextrakten der inaktivierten S-Stamm Pneumokokken Erbinformationen (konkret: die Erbinformationen zur Ausbildung der Schleimkapsel bzw. Virulenz) vorliegen müssen, die auf lebende R-Stamm Pneumokokken übertragen werden können und letztere in virulente S-Stamm Pneumokokken transformierten. Avery behandelte die Zellextrakte der virulenten S-Stamm Pneumokokken mit unterschiedlichen Enzymen, die gezielt spezifische Substanzen abbauen konnten. Darunter Protein abbauende Protease, Stärke abbauende Amylase, RNA abbauende RNase und DNA abbauende DNase. Nur die Behandlung des Zellextraktes mit DNase verhinderte die Transformation der R-Stamm Pneumokokken zur virulenten S-Stamm Form. Damit war bewiesen, dass die DNA die zur Ausbildung der Schleimkapsel und Virulenz erforderlichen Erbinformationen enthält.
Die Werkzeuge der Gentechnik: E. coli, Plasmide, Ligasen und Restriktionsenzyme
Im Jahre 1885 entdeckte der aus Ansbach stammende Kinderarzt Theodor Escherich ein zahlenmäßig häufig auftretendes und zur obligaten Normalflora des Dickdarms (Colon) gehörendes Bakterium, das er "Bacterium colon commune" nannte. Escherich erkannte, dass der Darm von Neugeborenen direkt nach der Geburt steril ist und sich die Besiedelung des Darms mit "gesunden Keimen", welche den Organismus vor schädlichen Keimen schützen, rasch nach dem ersten Stillen entwickelt. Er erkannte weiterhin, dass auch pathogene Varianten dieses Colonbakteriums existieren, die unter anderem als Erreger von Durchfallerkrankungen eine Rolle spielen. Zu Ehren des Entdeckers wurde das Bacterium colon commune später in Escherichia coli (kurz: E. coli) umbenannt. E. coli entwickelte sich in den folgenden Jahren zum Modellobjekt für die physiologische und pathogenitätsorientierte Körperfloraforschung, zum Indikator für die Verunreinigung von Gewässern und Lebensmitteln mit Fäkalien und zum beliebtesten "Versuchstierchen" der Molekulargenetiker, Biochemiker und später der Gentechniker.
Der Begriff "Plasmid" wurde 1952 von dem Molekularbiologen Joshua Lederberg als Bezeichnung für alle extrachromosomalen genetischen Partikel eingeführt. Plasmide sind natürlicherweise in Bakterien vorkommende, sich unabhängig vom Bakterienchromosom replizierende, zumeist ringförmige DNA-Moleküle. Als Werkzeuge der Gentechnik sind vor allem die so genannten Resistenzplasmide von großer Bedeutung, welche Gene zur Ausbildung von Resistenzen gegenüber Antibiotika oder anderen Toxinen enthalten und somit dem bakteriellen Träger einen Selektionsvorteil verschaffen. Mit der Zeit wurden Verfahren entwickelt, Plasmide gezielt aus Bakterien zu isolieren, diese außerhalb der Bakterienzelle zu verändern und anschließend wieder effizient in selbige einzuschleusen (Abbildung 3).
Die Ligase (von lateinisch ligare, verbinden) wurde im Jahre 1967 zeitgleich in mindestens vier verschiedenen Laboratorien unabhängig voneinander entdeckt. Dieses wichtige Werkzeug der Gentechnik besitzt die Eigenschaft, freie DNA-Enden miteinander zu verbinden. Da dieses Enzym ursprünglich aus einem E. coli-Stamm isoliert wurde, der mit dem T4-Bakteriophagen infiziert war, wird es auch als T4 DNA-Ligase bezeichnet (Abbildung 3).
Restriktionsenzyme sind Enzyme, die DNA-Doppelstränge an spezifischen Stellen aufschneiden können. Sie sind Teil einer bakteriellen "Immunabwehr" gegen Bakterien-befallende Viren, die Bakteriophagen. Die Bakterien besitzen dabei stets ein weiteres Enzym, die Methylase, welches ihre eigene DNA modifiziert (methyliert) und somit durch ein stammspezifisches Methylierungsmuster vor dem "Zerschneiden" durch die eigenen Restriktionsenzyme schützt. Nur Fremd-DNA, also z. B. die nicht-methylierte DNA von eingedrungenen Bakteriophagen, wird durch die eigenen Restriktionsenzyme gespalten (Abbildung 3). Das erste beschriebene Restriktionsenzym, EcoRI, wurde im Jahre 1969 von Werner Arber und Stuart Linn aus dem E. coli "R-Stamm" isoliert. Es schneidet nicht-methylierte, doppelsträngige DNA mit der spezifischen Nukleotidabfolge G-A-A-T-T-C.
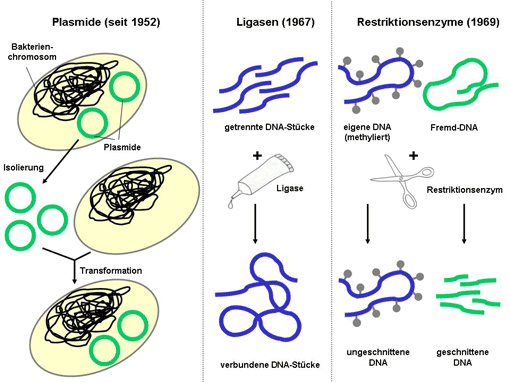
Abbildung 3: Die klassischen Werkzeuge der Gentechnik: Plasmide, Ligasen und Restriktionsenzyme. Plasmide sind natürlicherweise in Bakterien vorkommende, meist ringförmige "Zusatzchromosomen". Sie tragen häufig die genetische Information zur Ausbildung von Resistenzen gegenüber Antibiotika oder Toxinen und verschaffen somit dem bakteriellen Träger einen Selektionsvorteil. Plasmide können aus Bakterien isoliert, außerhalb der Bakterienzellen verändert und anschließend wieder in selbige eingeschleust (transformiert) werden. Die Ligase ist ein Enzym, das freie DNA-Enden miteinander verbinden kann. Restriktionsenzyme sind Teil der bakteriellen Immunantwort gegen Bakteriophagen und besitzen die Eigenschaft, DNA an spezifischen Stellen aufzuschneiden. Die eigene DNA der Bakterien wird dabei durch ein weiteres Enzym modifiziert (methyliert), so dass nur eingedrungene Fremd-DNA durch die Aktivität der Restriktionsenzyme zerstört wird.
Erste rekombinante DNA und erster gentechnisch veränderter Organismus
Im Jahre 1972, drei Jahre nach Entdeckung der Restriktionsenzyme, produzierte das Labor des Biochemikers Paul Berg an der Stanford Universität in Kalifornien (USA) das erste rekombinante DNA-Molekül. Rekombinante DNA bedeutet in diesem Zusammenhang eine im Labor (in vitro) erzeugte, natürlich nicht vorkommende Neukombination von DNA-Molekülen, die meist verschiedenen Organismen entstammen. Die von Paul Berg hergestellte rekombinante DNA bestand, vereinfacht ausgedrückt, aus zwei DNA-Molekülen, welche zuvor mit dem Restriktionsenzym EcoRI geschnitten und anschließend erneut mit Ligase zusammengefügt wurden. Ein Teilstück entstammte dabei aus dem Genom des Simian-Virus 40 (SV40), einem Affenvirus, welches unter bestimmten Umständen Tumore auslösen kann. Das zweite Fragment aus einem wenig charakterisierten E. coli Plasmid (Abbildung 4A).
Der erste gentechnisch veränderte Organismus wurde 1973 von Stanley Cohen et al. ebenfalls in Kalifornien (USA) generiert. Ein gentechnisch veränderter Organismus (abgekürzt: GVO) ist nach den Begriffsbestimmungen im Gentechnikgesetz ein Organismus, dessen genetisches Material in einer Weise verändert worden ist, wie es unter natürlichen Bedingungen durch Kreuzen oder natürliche Rekombination nicht vorkommt. Unter Organismus versteht man dabei jede biologische Einheit, die fähig ist, sich zu vermehren oder genetisches Material zu übertragen, einschließlich der Mikroorganismen, wie z. B. Bakterien, Viren oder Pilze. Die Herstellung des ersten GVO, einem gentechnisch veränderten E. coli, ist schematisch in der Abbildung 4B dargestellt. Hierfür wurden letztlich zwei natürlich vorkommende Plasmide mit unterschiedlichen Antibiotikaresistenzen mit dem Restriktionsenzym EcoRI linearisiert und danach mit Ligase zusammengefügt. Das neue Plasmid wurde in E. coli übertragen. Die gentechnisch veränderten Bakterien konnten anschließend durch Selektion mit beiden Antibiotika isoliert werden.
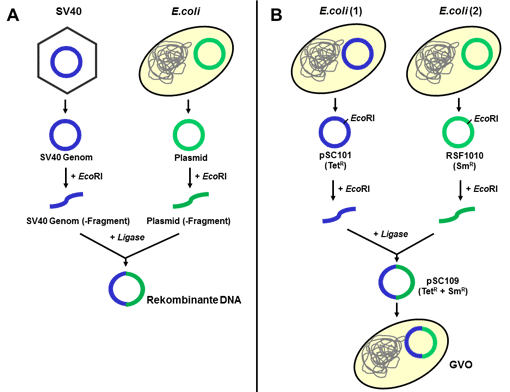
Abbildung 4. Erste rekombinante DNA (A) und erster gentechnisch veränderter Organismus (B). Zur Herstellung der rekombinanten DNA wurde das Genom des SV40 isoliert, mit EcoRI geschnitten und anschließend mit einem gleich behandelten, aus einem E. coli Stamm isolierten Plasmid zusammengefügt (A). Zur Herstellung des ersten GVO wurden die beiden natürlich in E. coli vorkommenden Resistenzplasmide pSC101 und RSF100 mit EcoRI linearisiert und anschließend ligiert. Das resultierende Plasmid pSC109 wurde dann in E. coli transformiert. Die gentechnisch veränderten E. coli Bakterien konnten durch Doppelselektion mit den Antibiotika Tetrazyklin und Streptomycin isoliert werden (B). Abkürzungen: SmR: Streptomycinresistenzgen, SV40: Simian-Virus 40, TetR: Tetrazyklinresistenzgen.
Von der ersten Asilomar Konferenz zum Gentechnikgesetz in Deutschland
Auf Initiative Paul Bergs trafen sich im Januar 1973 verschiedene nordamerikanische Wissenschaftler im kalifornischen Asilomar, um die potenziellen Risiken des gentechnischen Arbeitens mit Tumorviren, insbesondere SV40, zu diskutieren. Auslöser dieser "ersten Asilomar Konferenz" war das Vorhaben einer Mitarbeiterin Paul Bergs, die gesamte DNA von SV40 in E. coli einzuschleusen und die Befürchtung, ein Darmbakterium mit tumorigenen Eigenschaften zu erzeugen, welches beim Menschen z. B. Darmkrebs auslösen könnte. Auf der Konferenz wurde u. a. über persönliche Schutzmaßnahmen im Umgang mit GVO und spezifische Maßnahmen zur Einschließung von GVO diskutiert, um unbeabsichtigte Freisetzungen in die Umwelt zu verhindern.
Die Vorstellung weiterer neuer Methoden zur Herstellung rekombinanter DNA und zur artübergreifenden Übertragung von Genen auf der "Gordon Konferenz über Nukleinsäuren" im Juni 1973 löste eine erneute Sicherheitsdebatte unter den Wissenschaftlern aus. Zum Abschluss der Gordon Konferenz forderten die Teilnehmer die US-amerikanische Nationale Akademie der Wissenschaften (NAS) auf, eine offizielle Kommission mit der Untersuchung der potenziellen Risiken rekombinanter DNA einzusetzen.
Die entsprechende "Kommission über rekombinante DNA Moleküle" (englisch: Committee on Recombinant DNA Molecules) wurde noch im selben Jahr unter Vorsitz Paul Bergs, daher auch kurz "Berg-Kommission" genannt, gegründet. Im Juli 1974 veröffentlichte die Berg-Kommission zeitgleich einen offenen Brief im Publikationsorgan der US-amerikanischen Nationalen Akademie der Wissenschaften (Proceedings of the National Academy of Sciences of the United States of America), dem US-amerikanischen Science und dem britischen Nature Magazin.
Der offene Brief der Berg-Kommission enthielt folgende Forderungen:
- Freiwilliges, weltweites Moratorium von Experimenten mit rekombinanter DNA,
- Einberufung einer internationalen Experten-Konferenz zur Diskussion des weiteren Umgangs mit rekombinanter DNA,
- Einberufung einer Expertenkommission an den National Institutes of Health (NIH) zur Bewertung möglicher Sicherheitsrisiken beim Umgang mit rekombinanter DNA,
- Schaffung von Richtlinien für den Umgang mit rekombinanter DNA.
Im Februar 1975 fand eine internationale Experten-Konferenz im Rahmen der "Asilomar Konferenz über rekombinante DNA Moleküle" statt. Auf ihr diskutierten sowohl Wissenschaftler, als auch Juristen und Journalisten aus 17 verschiedenen Staaten Sicherheitskonzepte und Richtlinienvorschläge für den Umgang mit rekombinanter DNA. Zu nennen sind hier die Einführung eines Klassifizierungssystems zur Einteilung gentechnischer Arbeiten anhand ihres Gefährdungspotenzials für Mensch und Umwelt in vier Risikogruppen, die Einführung von (risikogruppenspezifischen) physikalischen und biologischen Sicherheitsmaßnahmen zur Verhinderung von unbeabsichtigten Freisetzungen in die Umwelt und das Einhalten einer guten Laborpraxis einschließlich der Unterweisung bzw. Schulung von Mitarbeitern. Als weiteres Resultat dieser Konferenz wurde das im Jahr zuvor ausgerufene, freiwillige Moratorium wieder aufgehoben. Weiterhin wurde ein Verbot bestimmter gentechnischer Arbeiten, z. B. mit hochpathogenen Mikroorganismen, mit sehr großen Kulturvolumen und aller Freisetzungen vereinbart.
Bereits 1974 wurde die Expertenkommission ("Recombinant DNA Advisory Committee" - RAC) an den National Institutes of Health (NIH), der nationalen Gesundheitsbehörde der USA, konstituiert. Als Konsequenz des bisherigen Prozesses und auch einer diesbezüglichen Anfrage der Weltgesundheitsorganisation (WHO) veröffentlichte das RAC im Juli 1976 die ersten so genannten "Richtlinien für die Forschung an rekombinanter DNA", auch kurz "NIH-Richtlinien" genannt. Das Dokument enthielt im Wesentlichen die bereits auf der zweiten Asilomar Konferenz diskutierten Richtlinienvorschläge. Weiterhin enthielt es konkrete Arbeitsanweisungen für bestimmte Arten von Experimenten mit rekombinanter DNAbzw.GVO, sowie eine Regelung von Zuständigkeiten und Verantwortlichkeiten (z. B. die Anzeige von Experimenten, das Erstellen von Hygieneplänen, die Nennung von Betreiber und Projektleiter, etc.).
Dem NIH-Konzept folgend wurde 1978 in Deutschland die "Zentrale Kommission für die Biologische Sicherheit" (ZKBS) am damaligen Bundesgesundheitsamt (BGA) eingerichtet und mit der Erstellung von "Richtlinien zum Schutz vor Gefahren durch in-vitro neukombinierte Nukleinsäuren", kurz "Genrichtlinien", beauftragt. Sie wurden noch im selben Jahr eingeführt. Ihre Inhalte beruhten zum Großteil auf den zwei Jahre zuvor veröffentlichten NIH-Richtlinien und waren für alle staatlichen Forschungseinrichtungen und öffentlich geförderten Projekte verbindlich. Von allen übrigen Einrichtungen wurde die Anwendung dieser Richtlinien auf freiwilliger Basis erwartet.
Im April 1990 erließ der Rat der Europäischen Wirtschaftsgemeinschaft (EWG) die beiden Richtlinien "90/219/EWG über die Anwendung genetisch veränderter Mikroorganismen in geschlossenen Systemen" (kurz: "Systemrichtlinie") und "90/220/EWG über die absichtliche Freisetzung von genetisch veränderten Organismen in die Umwelt" (kurz: "Freisetzungsrichtlinie"). Noch im Juni desselben Jahres verabschiedete der Deutsche Bundestag das erste "Gentechnikgesetz" (Gesetz zur Regelung der Gentechnik, GenTG). Die wesentlichen Leitgedanken des GenTG sind der Schutz von Mensch und Umwelt vor schädlichen Auswirkungen der Gentechnik sowie die Schaffung eines rechtlichen Rahmens für deren Erforschung, Entwicklung, Nutzung und Förderung.
In den Jahren seit 1990 wurden die ursprünglichen Fassungen der System- und Freisetzungsrichtlinie mehrfach geändert. Die derzeit aktuellen Fassungen sind die "Richtlinie 2009/41/EG" (Systemrichtlinie) und die "Richtlinie 2001/18/EG" (Freisetzungsrichtlinie). Auch das GenTG wurde seit seiner ersten Verabschiedung im Jahre 1990 mehrfach novelliert.
Die Abbildung 5 stellt chronologisch die wichtigsten internationalen und nationalen Entwicklungen seit 1972 dar, die zur Entstehung des GenTGin Deutschland führten.
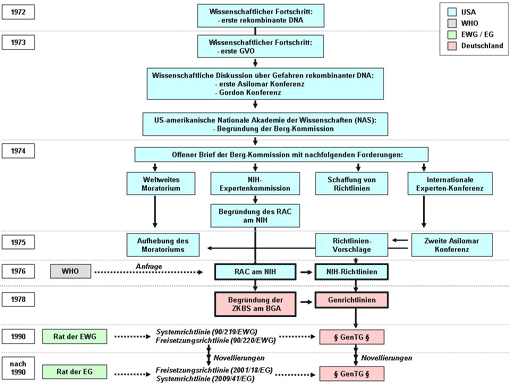
Abbildung 5: Chronologie der wichtigsten internationalen und nationalen Entwicklungen seit 1972 die zur Entstehung des GenTG in Deutschland führten. Erklärung siehe Text. Abkürzungen: BGA: Bundesgesundheitsamt, EG: Europäische Gemeinschaft, EWG: Europäische Wirtschaftsgemeinschaft, GenTG: Gesetz zur Regelung der Gentechnik (Gentechnikgesetz), GVO: gentechnisch veränderte Organismen, NIH: National Institutes of Health, NAS: National Academy of Sciences, RAC: Recombinant DNA Advisory Committee, USA: United States of America, WHO: World Health Organization, ZKBS: Zentrale Kommission für die Biologische Sicherheit.
Mehr zu diesem Thema
Allgemeine Informationen zum Thema
Gentechnische Arbeiten in gentechnischen Anlagen
- Gentechnische Anlagen: Behörden und Institutionen
- Gentechnische Arbeiten
- Gentechnische Anlagen
- Betreiber, Projektleiter und Beauftragte für die Biologische Sicherheit
- Verwaltungsverfahren für gentechnische Anlagen und gentechnische Arbeiten
- Tätigkeiten des LGL im Rahmen der Überwachung gentechnischer Anlagen
- Gentechnische Anlagen: Häufig gestellte Fragen - Frequently asked questions (FAQ)
- Genttechnische Anlagen: Weiterführende Links

