- Startseite >>
- Gesundheit >>
- Infektionsschutz >>
- Molekulare Surveillance >>
- BIS+C
Virale Atemwegserreger in Bayern - Auswertung 2024/2025
Abstract
Das Bayern Influenza + Corona Sentinel (BIS+C) bildet gemeinsam mit dem Bay-VOC Projekt und dem Abwassermonitoring die drei Säulen der molekularen Überwachung viraler Atemwegserreger in Bayern. Im BIS+C werden stichprobenartig Nasen- bzw. Rachenabstriche von Patientinnen und Patienten mit Symptomen einer akuten Atemwegserkrankung (= akute respiratorische Erkrankung, ARE) aus bayerischen Hausarzt-, Kinder- und Jugendarztpraxen, den Sentinelpraxen, an das Bayerische Landesamt für Gesundheit und Lebensmittelsicherheit (LGL) gesandt. Die Proben werden labordiagnostisch auf Influenza-, SARS-CoV-2 und Respiratorische Synzytial-Viren (RSV) sowie deren Varianten untersucht. Durch die Projektförderung konnte das Netzwerk erfolgreich ausgebaut und erhalten werden: Im Jahr 2024/25 nahmen rund 200 Praxen am Sentinel teil. Die gewonnenen epidemiologischen Daten ermöglichen eine Überwachung viraler Atemwegserreger sowie die zeitnahe Erkennung von Trends.
Hintergrund
Das BIS existiert bereits seit 2009 und dient der virologischen Überwachung von akuten Atemwegserkrankungen im Freistaat Bayern. Der ursprüngliche Fokus lag auf Influenza- und RSV, wobei letztere damals nur bei Kindern unter fünf Jahren überwacht wurden. Im Mai 2022 beschloss die Bayerische Staatsregierung die Förderung und den Ausbau des BIS. Im Zuge dessen wurde das Sentinel auf die Untersuchung von RSV und SARS-CoV-2 einschließlich ihrer Virusvarianten in allen Altersgruppen ausgeweitet und in BIS+C umbenannt. Die SARS-CoV-2-positiven Proben aus den Sentinelpraxen werden zusätzlich mittels DNA-Sequenzierung, genauer Next Generation Sequencing, analysiert. Die gewonnenen genetischen Informationen fließen in das molekulargenetische Überwachungsnetzwerk Bay-VOC ein.
Nur durch eine kontinuierliche Surveillance können Änderungen im Verteilungsmuster viraler Atemwegserreger in Bayern unmittelbar erkannt und damit die Bevölkerung besser informiert und geschützt werden. Auf Basis dieser Daten können Impfkampagnen gegen Influenza-, RSV- und SARS-CoV-2 gezielt geplant und rechtzeitig umgesetzt werden. Dies ermöglicht den Praxen der Primärversorgung eine vorausschauende Impfstoffbestellung und -bevorratung, was zur Reduktion schwerer Krankheitsverläufe beiträgt. Krankenhäuser können sich rechtzeitig auf einen hohen Patientenandrang vorbereiten und frühzeitig vorbeugende Maßnahmen ergreifen.
Ergebnisse
Seit 2022 senden niedergelassene Arztpraxen mit einer pädiatrischen oder Hausarztfunktion im Rahmen des BIS+C ganzjährig wöchentlich vier bis sechs Nasen- bzw. Rachenabstriche von Patienten mit akuter Atemwegserkrankung-Symptomatik an das LGL. Das Sentinel-Netzwerk wurde in Zusammenarbeit mit dem Bayerischen Staatsministerium für Gesundheit, Pflege und Prävention (StMGP), dem Bayerischen Hausärzteverband, dem bayerischen Berufsverband der Kinder- und Jugendärzte e. V. sowie der Kassenärztlichen Vereinigung Bayerns (KVB) erfolgreich ausgebaut. Die Zahl der teilnehmenden Sentinelpraxen stieg von 92 (Saison 2021/22) auf 205 (Stand KW 06/2025). Diese Ausweitung führte zu einer deutlichen Steigerung der Probenzahl und damit zu einer verbesserten Aussagefähigkeit über die Situation viraler Atemwegserkrankungen in den Arztpraxen der bayerischen Primärversorgung.
Die wöchentliche Auswertung der ARE-Daten umfasst neben der Typisierung der zirkulierenden Influenza-, RSV- und SARS-CoV-2-Viren dabei auch die Dokumentation der Krankheitssymptome sowie die Bestimmung von Beginn, Verlauf und Höhepunkt der jeweiligen Viruswellen anhand der Positivenraten pro Kalenderwoche. Die Daten werden in wöchentlichen BIS+C-Berichten auf der LGL-Homepage veröffentlicht und zeigen den Verlauf des Infektionsgeschehens in Bayern auf.
Auf diese Weise können Trends und Unterschiede zu Vorjahren frühzeitig entdeckt und im Bedarfsfall rasch Maßnahmen ergriffen werden. Zudem fließen die Daten SARS-CoV-2 positiver Abstriche in den Bay-VOC-Bericht und die Bay-VOC Homepage, die nationale sowie internationale Überwachung der Influenza- und SARS-CoV-2-Viren sowie in die Impfstoffempfehlungen der Weltgesundheitsorganisation ein. Die Ergebnisse aus dem BIS+C Vorjahr 2023/2024 sind im detaillierten Abschlussbericht (PDF, 500 KB) dokumentiert. Die aktuellen Ergebnisse des BIS+C Jahres 2024/2025 (KW 40/2024 bis Juni 2025) werden in den Abbildungen 1 bis 5 dargestellt.
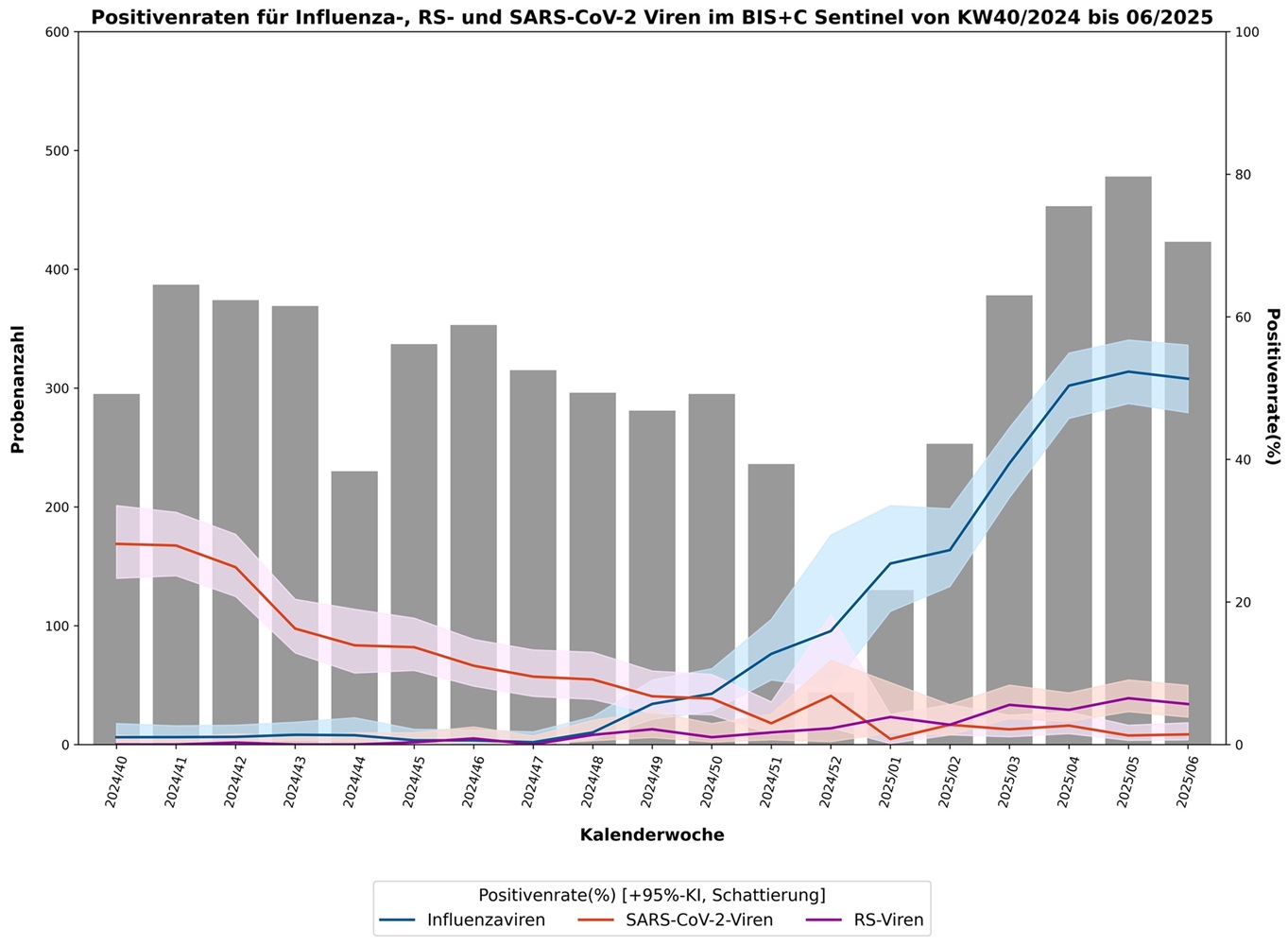
Abbildung 1: Positivenraten für Influenza-, SARS-CoV-2 und RSV der erhaltenen Nasen- bzw. Rachenabstrichproben im BIS+C Sentinel von KW 40/2024 bis KW 6/2025. Es zeigt sich, dass SARS-CoV-2 das virale Infektionsgeschehen der drei Erreger bis KW 40/2024 dominierte und seitdem die Positivenrate stetig sinkt. Seit KW 01/2025 wurde nach Definition des Robert Koch-Instituts (RKI) die Grippewelle erklärt, welche dieses Jahr vor allem durch die beiden Erreger A(H1N1)pdm09 sowie Influenza B hervorgerufen wird. Nach RKI-Definition trat ab KW 03/2025 im BIS+C Sentinel zudem die RSV-Welle in Bayern auf, welche vor allem die 0 bis 4-Jährigen betraf.
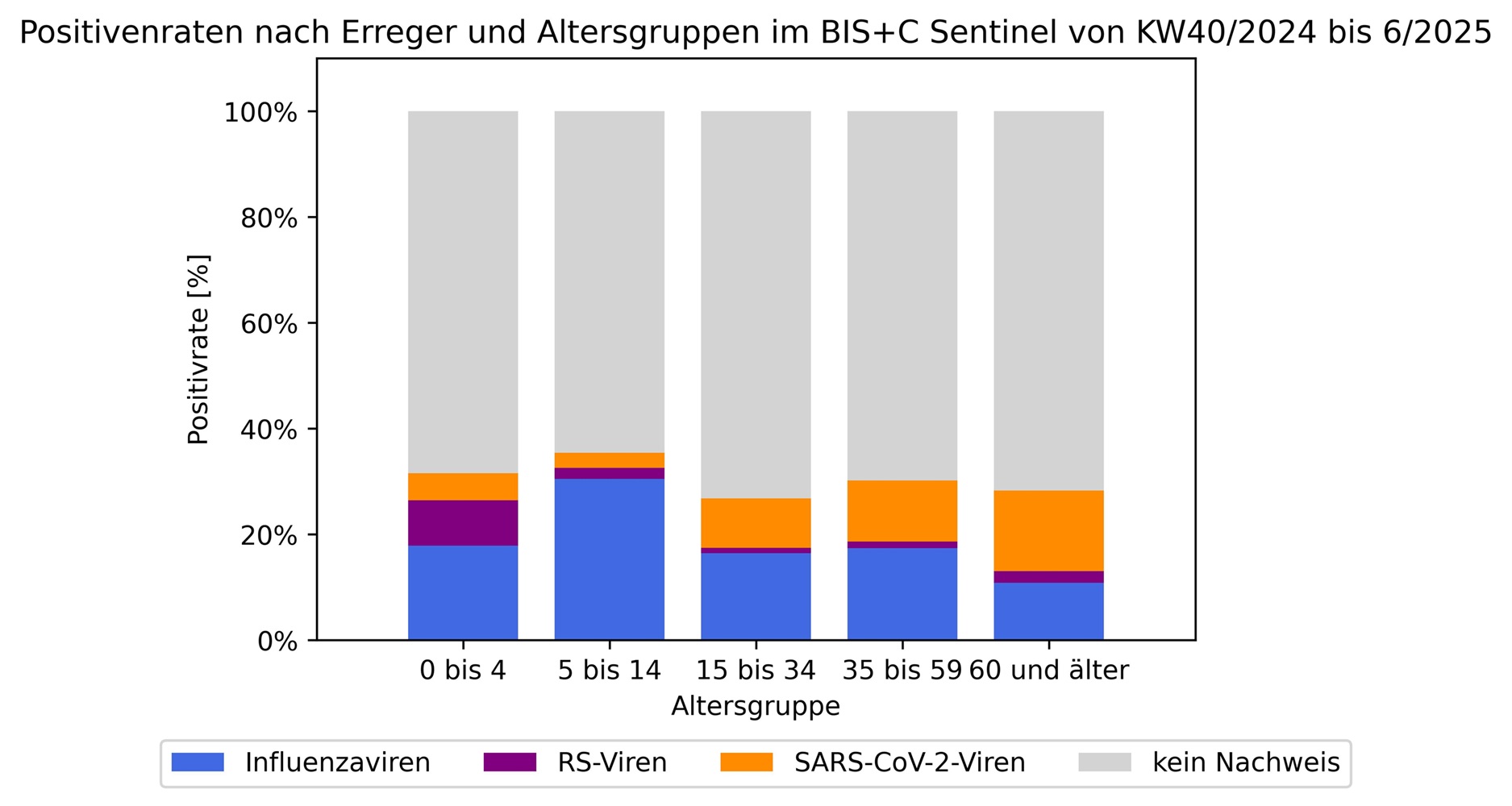
Abbildung 2: Positivenraten für Influenza-, SARS-CoV-2- und RSV der erhaltenen BIS+C Abstriche von KW 40/2024 bis KW 06/2025 nach Altersgruppen. Es ist zu erkennen, dass im genannten Zeitraum RSV vor allem Kinder der Altersgruppe 0 bis 4 Jahre gefolgt von der Altersgruppe der 5- bis 14-Jährigen und der Altersgruppe der Senioren über 60 Jahre betraf. SARS-CoV-2 betraf insbesondere die Altersgruppe der Senioren über 60 Jahre sowie Erwachsene ab 35 bis 59 Jahre als auch Jugendliche ab 15 Jahren. Influenza-Fälle traten in allen Altersgruppen auf, aber vor allem die Altersgruppe der 5- bis 14-Jährigen war von der aktuellen Grippewelle betroffen.
Abbildung 3: Verteilung der Influenzavirus-Subtypen im BIS+C Sentinel von KW 40/2024 bis KW 06/2025. Der Großteil der insgesamt 1.134 Influenza Infektionen der eingegangenen 6.083 Abstriche war auf zwei Erreger zurückzuführen: Influenza B (46,8%) und Influenza A(H1N1)pdm09 (41,6%) gefolgt vom Erreger Influenza A(H3N2) (8,4%). Zudem gab es einige Influenza A (3,2%) Infektionen, die (noch) nicht subtypisierbar waren. Insgesamt traten fünf Koinfektionen mit Influenza A und B Viren auf.
SARS-CoV-2 positiv getestete Proben aus dem BIS+C Sentinel mit ausreichend hoher Viruslast fließen bereits seit dem Jahr 2021 in die Ganzgenomsequenzierung (Sequenzierung des kompletten genetischen Erbguts) im Rahmen des mit BIS+C assoziierten Bay-VOC Projektes ein. Anhand der Ganzgenomdaten können so die vorhandenen Viruslinien im Detail identifiziert werden. Die Linien unterscheiden sich durch ihr jeweiliges Mutationsprofil, das die Infektionsrate und damit die Verbreitung und Prävalenz der jeweiligen Linie im Vergleich zu anderen Linien beeinflusst.
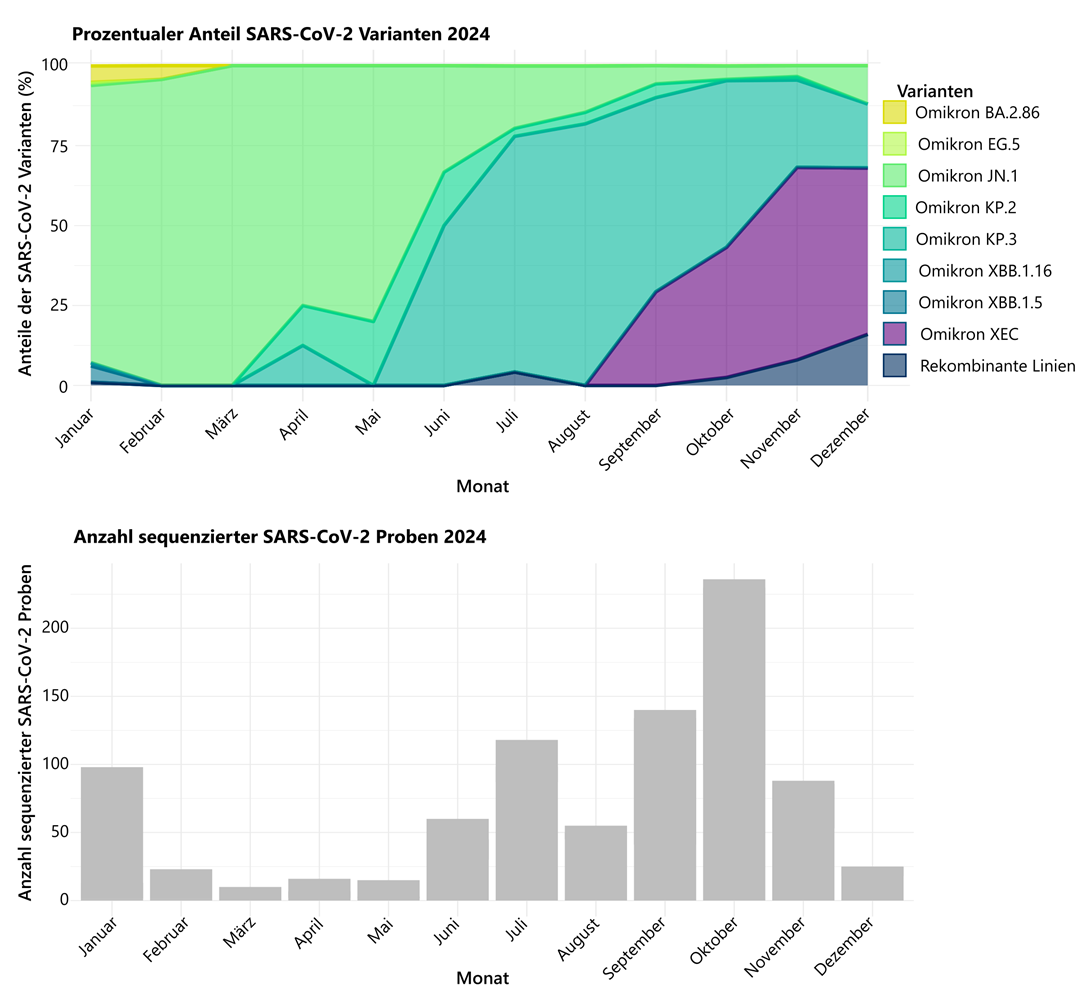
Im Verlauf des Jahres 2024 wurden in 980 SARS-CoV-2 positiven BIS+C Proben verschiedene Sublinien der von der Weltgesundheitsorganisation WHO definierten „Variant of Concern“ (VOC) Omikron identifiziert, wobei sich die prozentuale Linienzusammensetzung im untersuchten Probenkollektiv im Jahresverlauf aufgrund der fortschreitenden Virusevolution und das damit zusammenhängende Auftreten und die Prävalenzzunahme neuer Sublinien immer wieder änderte:
- Beginn des Jahres 2024: Die dominierende Linie war zunächst JN.1, die aus der Omikron-Linie BA.2.86 hervorgegangen war, die bereits im Jahr 2023 eine Rolle gespielt hatte. JN.1 hatte Anfang 2024 eine Prävalenz von über 90 %, wurde aber ab März langsam abgelöst, insbesondere durch die aus ihr hervorgegangene Sublinie KP.3, die fast zeitgleich mit der Linie KP.2 auftrat.
- Sommer bis Jahresende 2024: KP.2 blieb prozentual auf niedrigem Niveau, während KP.3 zur dominierenden Linie wurde und im Sommer eine Prävalenz von über 80 % erreichte. Linie KP.3 wurde als dominante Linie ab August durch die rekombinante Linie XEC abgelöst, die aus zwei Vorgängerlinien hervorgegangen ist, die wiederum von JN.1 abstammten. Sie erreichte bis zum Jahresende eine Prävalenz von ca. 80 % (siehe Abb. 5).
Um die aktuell zirkulierenden Viruslinien und ihren Verlauf übersichtlich öffentlich zugänglich zu machen, werden diese Daten, ebenso wie die Viruslinien der parallel durchgeführten Ganzgenomsequenzierungen aus dem bayerischen Abwassermonitoring, kontinuierlich und in interaktiver Form auf der Bay-VOC Website dargestellt. Abbildung 4 zeigt detailliert die Ergebnisse des Bay-VOC-Projekts für alle SARS-CoV-2 positiven BIS+C Proben, die im Jahr 2024 am LGL analysiert wurden.
Für Influenza A positive Patientenproben des BIS+C Sentinels wurde 2024 im Rahmen des assoziierten Bay-VOC-Projekts am LGL ebenfalls ein Workflow zur Ganzgenomsequenzierung etabliert. Dieser wird ab dem Jahr 2025 sukzessive in die Routine überführt, um im BIS+C Sentinel auch innerhalb der Influenza A-Subtypen Viruslinien detailliert zu erfassen.
Abbildung 4: SARS-CoV-2 Viruslinien im Jahr 2024: Prozentuale Verteilung der nachgewiesenen Varianten (oben) und Anzahl der sequenzierten SARS-CoV-2 positiven Proben pro Monat (unten). Berücksichtigt wurden nur BIS+C-Proben mit für die Genomsequenzierung ausreichender Viruslast.
Abbildung 5: Probenanzahl und Positivenraten für SARS-CoV-2, Influenza und RSV im Vergleich der BIS+C-Jahre 2022/23 bis 2024/25 (Stand KW 06/2025):
Die grauen Balken zeigen die Gesamtzahl der untersuchten Proben, die Linien mit Schattierungen zeigen die jeweiligen Positivenraten mit 95%-Konfidenzintervallen. Der erfolgreiche Ausbau des Sentinels zeigt sich in den gestiegenen Probenzahlen: von über 10.000 Proben in 2022/23 auf 13.629 Proben in 2023/24. Im laufenden BIS+C-Jahr 2024/25 wurden bereits 6.083 Proben untersucht. Auffällig ist die zeitliche Verlagerung der SARS-CoV-2-Positivenraten in Phasen ohne Influenza- und RSV-Aktivität.
Fazit
Das von der Bayerischen Staatsregierung geförderte und ausgebaute BIS+C Projekt am LGL hat sich als effektives Instrument der molekularen und virologischen ARE-Surveillance in Bayern etabliert. Es erfasst systematisch und stichprobenartig die Positivenraten von Influenza-, SARS-CoV-2 und RSV-Infektionen von Patienten mit Atemwegserkrankungen in bayerischen Arztpraxen der Primärversorgung und dokumentiert deren Krankheitswellen in der Bevölkerung. Gemeinsam mit dem Bay-VOC Projekt und dem Abwassermonitoring bildet das BIS+C Projekt die drei Säulen der molekularen Surveillance viraler ARE-Erreger im Freistaat Bayern.
Diese drei Säulen ermöglichen nicht nur einen Einblick in das virale Infektionsgeschehen von SARS-CoV-2, Influenza und RSV innerhalb der von Erkrankten gut frequentierten, bayerischen Arztpraxen der Primärversorgung, sondern überblicken dank des Abwassermonitorings und seiner Vielzahl von Standorten auch einen großen Teil der bayerischen Bevölkerung – auch für jene Bürger, die nicht zum Arzt gehen, asymptomatisch erkrankt oder sogar gesund sind. Nur so kann der Ist-Zustand an respiratorischen Viruserkrankungen innerhalb der bayerischen Bevölkerung möglichst genau ermittelt und eine verlässliche Aussage über die Infektionslast viraler Atemwegserreger in Bayern getroffen werden. Dank des Bay-VOC Projekts konnte von allen aus dem BIS+C Sentinel und dem Abwassermonitoring gewonnenen SARS-CoV-2 positiven Proben mit ausreichend hoher Viruslast ein molekularer Fingerabdruck mittels Next Generation Sequencing gewonnen werden, der die in Bayern im Jahr 2024 kursierenden SARS-CoV-2 Varianten detailgenau im Kontext der globalen SARS-CoV-2 Evolution widerspiegelt.
Der erfolgreiche Aufbau des BIS+C-Sentinels hat so eine wichtige, verzahnte Surveillance-Struktur geschaffen, deren Erhalt für die weitere ARE-Überwachung in Bayern von großer Bedeutung ist. Für 2025 ist geplant, das Spektrum der überwachten Erreger zusätzlich zu erweitern und Influenza-A-positive Proben im Rahmen des Bay-VOC-Projekts zu sequenzieren. Nur durch den Erhalt der drei Säulen der molekularen Surveillance viraler ARE-Erreger in Bayern ist gewährleistet, dass sowohl virale Erreger kranker als auch gesunder Personen gleichermaßen molekular analysiert und möglichst frühzeitig dargestellt werden.
Literatur
- Cambridge University Press: Reciprocal circulation pattern of SARS-CoV-2 and influenza viruses during the influenza seasons 2019/2020 and 2020/2021 in the Bavarian Influenza Sentinel (Germany) (https://www.cambridge.org/core/journals/epidemiology-and-infection/article/reciprocal-circulation-pattern-of-sarscov2-and-influenza-viruses-during-the-influenza-seasons-20192020-and-20202021-in-the-bavarian-influenza-sentinel-germany/569E0C4EB9940C1C7DFE537470585425)
- Vergleich der SARS-CoV-2-und Influenzavirusinfektionen (https://www.bayerisches-aerzteblatt.de/fileadmin/aerzteblatt/ausgaben/2021/06/einzelpdf/BAB_6_2021_278_279.pdf)
- Bericht zur Influenzaüberwachung in Bayern 2018/19 (https://www.bayerisches-aerzteblatt.de/fileadmin/aerzteblatt/ausgaben/2019/11/einzelpdf/596-598.pdf)
- Die Influenzasaison 2016/17 in Bayern (https://www.lgl.bayern.de/downloads/gesundheit/infektionsschutz/doc/bab_influenza_2016_2017.pdf)
- Bayern Influenza Sentinel (BIS) der Influenzasaison 2016/17 (https://www.lgl.bayern.de/downloads/gesundheit/infektionsschutz/doc/bis_daten_2016_2017.pdf)
- Erregermonitoring im Bayern Influenza Sentinel (EMBIS) in Kalenderwochen 7 bis 16 2014 (https://www.lgl.bayern.de/downloads/gesundheit/infektionsschutz/doc/embis_daten_2014.pdf)
- Eberle, U., Heinzinger, S., Konrad, R. et al. Virological COVID-19 surveillance in Bavaria, Germany suggests no SARS-CoV-2 spread prior to the first German case in January 2020. Infection 49, 1029–1032 (2021). (https://doi.org/10.1007/s15010-021-01611-y)
- Flechsler, J., Eberle, U., Dangel, A. et al. Molecular SARS-CoV-2 surveillance in Bavaria shows no Omicron transmission before the end of November 2021. Infection 50, 761–766 (2022). (https://doi.org/10.1007/s15010-022-01767-1)
- Jungnick, S.; Hobmaier, B.; Mautner, L. et al. In Vitro Rapid Antigen Test Performance with the SARS-CoV-2 Variants of Concern B.1.1.7 (Alpha), B.1.351 (Beta), P.1 (Gamma), and B.1.617.2 (Delta). Microorganisms 2021, 9, 1967. (https://doi.org/10.3390/microorganisms9091967)
- Jungnick S., Hobmaier B., Mautner L. et al. Detection of the new SARS-CoV-2 variants of concern B.1.1.7 and B.1.351 in five SARS-CoV-2 rapid antigen tests (RATs), Germany, March 2021. Euro Surveill. 2021;26(16):pii=2100413. (https://doi.org/10.2807/1560-7917.ES.2021.26.16.2100413)
- Jungnick, S., Hobmaier, B., Paravinja, N. et al. Analysis of seven SARS-CoV-2 rapid antigen tests in detecting omicron (B.1.1.529) versus delta (B.1.617.2) using cell culture supernatants and clinical specimens. Infection 51, 239–245 (2023). (https://doi.org/10.1007/s15010-022-01844-5)
Mehr zu diesem Thema
Allgemeine Informationen zum Thema
Weitere LGL-Artikel
- Neuartiges Coronavirus SARS-CoV-2 (COVID-19)
- Epidemiologische Berichterstattung und Surveillance-Daten
- BIS+C Sentinel-Arztpraxen
- Daten zur Grippesaison in Bayern – Influenza-Meldungen nach Infektionsschutzgesetz (IfSG)